Nguồn Gốc Kỹ Thuật
Real-time PCR là gì?
Trong kỹ thuật PCR, sau khi hoàn tất khuếch đại đoạn DNA đích, người làm thí nghiệm phải tiếp tục làm một số bước thí nghiệm để đọc kết quả xác định có sản phẩm khuếch đại mong muốn trong ống phản ứng hay không, và giai đoạn này gọi là giai đoạn thí nghiệm sau PCR. Trong giai đoạn này, người làm thí nghiệm có thể thực hiện điện di sản phẩm PCR trên gel agarose để xem có vạch sản phẩm khuếch đại đúng kích thước mong muốn hay không, cũng có thể thực hiện thí nghiệm lai với các đoạn dò đặc hiệu (trên màng, trên giếng hay phiến nhựa…) để xem sản phẩm khuếch đại có trình tự mong muốn hay không. Kỹ thuật PCR mà cần phải có giai đoạn thí nghiệm để đọc và phân tích sau khi hoàn tất phản ứng khuếch đại, ngày hôm nay được gọi là PCR cổ điển (classical PCR).
Real-time PCR là kỹ thuật PCR mà kết quả khuếch đại DNA đích hiển thị được ngay sau mỗi chu kỳ nhiệt của phản ứng, chính vì vậy nên được gọi là real-time; và do đặc điểm này nên với real-time PCR người làm thí nghiệm không cần thiết phải làm tiếp các thí nghiệm để đọc và phân tích kết quả để xác định có sản phẩm khuếch đại đích hay không vì kết quả cuối cùng của phản ứng khuếch đại cũng được hiển thị ngay sau khi hoàn tất phản ứng khuếch đại. Như vậy, nên có thể nói real-time PCR là kỹ thuật nhân bản DNA đích trong ống nghiệm thành hàng tỷ bản sao dựa vào các chu kỳ nhiệt và kết quả khuếch đại trong ống phản ứng được hiển thị cùng lúc với phản ứng khuếch đại xảy ra để người làm thí nghiệm có thể thấy được.
Các vấn đề kỹ thuật cơ bản cần biết của real-time PCR
1. Biểu đồ khuếch đại của real-time PCR
Trong real-time PCR, hiển thị cơ bản để người làm thí nghiệm có thể quan sát được trong quá trình nhân bản DNA của các ống phản ứng là một biểu đồ khuếch đại (amplification graph). Biểu đồ này có trục tung (Y) là cường độ huỳnh quang phát ra từ các ống phản ứng khi nhận ánh sáng kích thích, còn trục hoành (X) là các chu kỳ nhiệt. Trên biểu đồ khuếch đại này (biểu đồ 1), người làm thí nghiệm sẽ thấy đối với từng
ống phản ứng, cường độ huỳnh quang mà máy ghi nhận được trong những chu kỳ đầu sẽ rất thấp và hầu như không thay đổi, hiển thị bằng một đường thẳng nằm ngang, chúng ta có thể gọi đây là “giai đoạn ủ” hay “giai đoạn tiềm phục”, vì trong giai đoạn này dù DNA đích đã có thể được nhân bản thành các bản sao nhưng do số lượng chưa đủ để giúp cho chất phát huỳnh quang nhận được ánh sáng kích thích phát ra ánh sáng huỳnh quang đủ cường độ để máy ghi nhận. Nhưng một khi số lượng bản sao của DNA đích đạt đến một ngưỡng nhất định thì ánh sáng huỳnh quang phát ra sẽ đủ cường độ để được máy ghi nhận và lúc này chúng ta sẽ thấy đường biểu diễn khuếch đại bắt đầu ngóc lên. Cường độ huỳnh quang trong ống phản ứng từ lúc này trở đi sẽ tăng gấp đôi sau mỗi chu kỳ nhiệt do số lượng bản sao của DNA đích tăng gấp đôi sau mỗi chu kỳ. Chúng ta gọi giai đoạn này là “giai đoạn lũy thừa” về cường độ huỳnh quang, không phải là giai đoạn tăng trưởng lũy thừa về số lượng bản sao của DNA đích. Cường độ huỳnh quang trong ống phản ứng sẽ tăng trưởng đến một mức nào đó thì độ tăng trưởng sẽ chậm dần và đạt đến bình nguyên vì các bản sao của DNA đích, do phản ứng đã cạn dần dNTP và enzyme Taq polymerase không hoạt động hiệu quả nữa, nên sẽ không còn gia tăng số lượng theo cấp số 2 nữa. Chúng ta gọi giai đoạn này là “giai đoạn bình nguyên”.
Phân tích một đường biểu diễn khuếch đại (amplification curve) của một ống phản ứng sau khi hoàn tất được các chu kỳ nhiệt, chúng ta sẽ thấy một thông số hết sức quan trọng luôn đi kèm với nó, đó là chu kỳ ngưỡng (Ct, threshold cycle). Chu kỳ ngưỡng hay Ct là chu kỳ nhiệt mà ở tại thời điểm này thiết bị real-time ghi nhận được tín hiệu huỳnh quang phát ra từ ống phản ứng bắt đầu vượt qua cường độ huỳnh quang nền. Để có thể xác định được cường độ huỳnh quang nền, thiết bị real-time thường ghi nhận cường độ tín hiệu huỳnh quang xuất hiện trong ống phản ứng trong một số chu kỳ đầu, chúng ta gọi là các chu kỳ nền (basal cycle), và lấy trung bình cộng của các cường độ huỳnh quang này làm cường độ huỳnh quang nền. Đường cắt ngang đi qua cường độ huỳnh quang nền này được gọi là đường nền (base line). Chu kỳ ngưỡng là trị số được xác định bằng số chu kỳ mà ở đó đường nền cắt được đường biển diễn khuếch đại. Do được tính toán như vậy nên chu kỳ ngưỡng thường là một số lẻ (ví dụ: Ct = 28.35) chứ ít khi là một số chẵn.
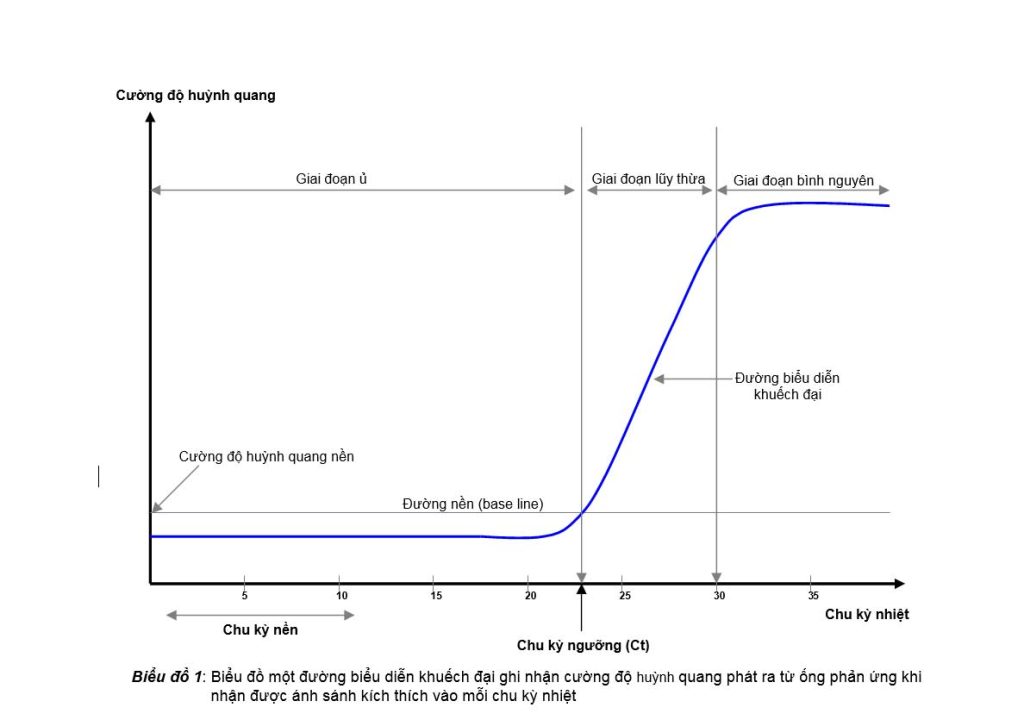
Có những ống phản ứng có Ct sớm và cũng có những ống phản ứng có Ct xuất hiện muộn hơn, và câu hỏi được đặt ra là lý do nào đã làm được như vậy? Câu trả lời chính xác là do số lượng bản DNA đích ban đầu (starting quantity, Sq) có trong ống phản ứng nhiều hay ít. Nếu trong ống phản ứng số lượng DNA đích nhiều thì sẽ cần ít chu kỳ nhiệt hơn để đạt đến số lượng bản sao đủ để ống phản ứng cho được tín hiệu huỳnh quang mà máy sẽ ghi nhận được, còn nếu số lượng DNA đích ít hơn thì cần nhiều chu kỳ nhiệt hơn.
2. Biểu đồ chuẩn của real-time PCR
Như đã nói Ct của một ống phản ứng real-time PCR sớm hay muộn (Ct thấp hay Ct cao) là tùy thuộc vào số lượng bản DNA đích ban đầu có trong ống phản ứng. Đây chính là một đặc điểm vượt trội của real-time PCR so với PCR cổ điển, vì nhờ dựa vào đặc điểm này mà người làm thí nghiệm xác định được số copies của tác nhân đích có trong mẫu thử, một thông số mà PCR cổ điển không thể nào làm để có được kết quả chính xác. Người làm thí nghiệm chỉ đọc kết quả PCR cổ điển sau khi hoàn tất phản ứng khuếch đại, và kết quả này nếu định lượng được thì cũng chỉ là kết quả định lượng
số bản sao của DNA đích sau khi hoàn tất khuếch đại. Mà trong PCR, số lượng bản sao cuối cùng không phản ảnh được một cách chính xác số lượng bản DNA đích ban đầu có trong mẫu thử vì trong đa số các trường hợp số lượng bản sao có trong
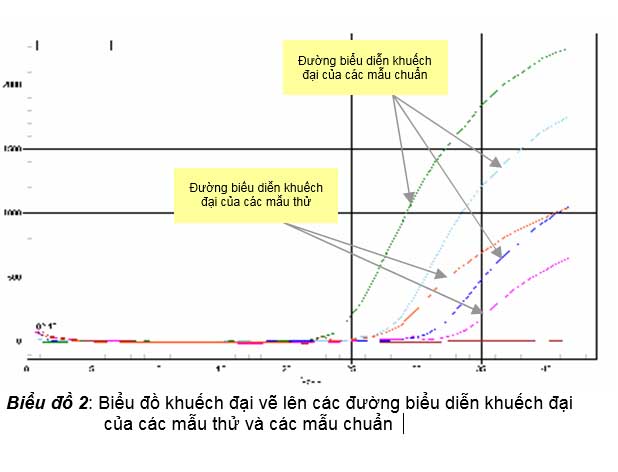
ống phản ứng PCR là số lượng cực đại sau khi PCR đạt được giai đoạn bình nguyên.
Tuy nhiên để real-time PCR xác định được chính xác số lượng bản đích ban đầu có trong mẫu thử, người làm thí nghiệm phải thực hiện real-time PCR các mẫu thử chứa các số lượng bản DNA đích ban đầu cần xác định số lượng cùng lúc với các mẫu chuẩn chứa số lượng DNA đích ban đầu đã biết rõ số lượng, và thường thì các mẫu chuẩn là các mẫu pha loãng theo hệ số pha loãng 10 chứa số lượng DNA đích ban đầu. Sau khi hoàn tất các chu kỳ nhiệt của real-time PCR, trên biểu đồ khuếch đại (biểu đồ 2), các mẫu chuẩn và các mẫu thử sẽ có các đường biểu diễn khuếch đại của nó và tương ứng với các đường biểu diễn
khuếch đại này, các mẫu chuẩn và mẫu thử đều có một chu kỳ ngưỡng (Ct).
Đường biểu diễn vẽ lên mối quan hệ giữa chu kỳ ngưỡng với số lượng bản DNA đích ban đầu có trong các mẫu chuẩn được gọi là đường biểu diễn chuẩn (standard
curve). Đây là một đường thẳng
tuyến tính đi qua các điểm tọa độ xác định bởi số lượng bản DNA đích ban đầu của từng mẫu chuẩn
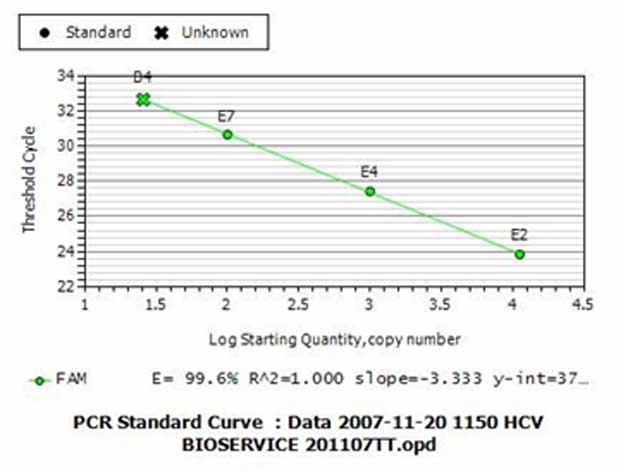
Biểu đồ 3: Biểu đồ chuẩn vẽ lên đường biểu diễn chuẩn về mối quan hệ giữa số lượng bản DNA đích có trong các mẫu chuẩn và chu kỳ ngưỡng tương ứng. Trong biểu đồ ở trên này, chúng ta E2-E4-E7 là các ống phản ứng chứa các mẫu chuẩn còn B4 là ống phản ứng chứa mẫu thử.
(trên trục tung) và chu kỳ ngưỡng của ống phản ứng chứa số lượng bản DNA đích này (biểu đồ 3). Trên biểu đồ chuẩn này, trục tung (Y) là Ct còn trục hoành (X) là số lượng bản DNA đích ban đầu có trong các mẫu chuẩn. Do các mẫu chuẩn thường được pha cách nhau theo hệ số pha loãng 10, nên số lượng bản DNA đích trong các mẫu chuẩn được biểu thị bằng logarith cơ số 10 (log10) của số lượng này. Do vậy trị 1.5, 2, 2.5, 3, 3.5, 4, 4.5 trên trục X là tương ứng với các số lượng bản đích là 101.5, 102, 102.5, 103,
103.5, 104, 104.5 tức là 32, 100, 316, 1000, 3162, 10000, 31623 copies trong ống phản
ứng.
Có hai thông số được máy tính toán và hiển thị trên một đường biểu diễn chuẩn. Hai thông số này rất có giá trị để người làm thử nghiệm có thể đánh giá được thao tác pipetting của mình trong quá trình làm thử nghiệm.
Thông số đầu tiên là hệ số tương quan R2, đánh giá độ chính xác của thao tác pipetting lấy có đúng thể tích mong muốn hay không. Trị số R2 phải đạt là trên hay bằng (≥) 0.99 có nghĩa là đường biểu diễn chuẩn phải đạt tuyến tính cao. Khi người làm thử nghiệm không lấy được các thể tích chính xác thì chắc chắn các thể tích mẫu chuẩn cho vào real-time PCR mix sẽ không thể nào chính xác được, nên R2 chắc chắn sẽ không thể nào đạt ≥ 0.99.
Một trị số nữa rất có giá trị mà người làm thử nghiệm phải biết rõ đó là hiệu quả PCR, được gọi là E% (PCR efficiency). Một khi PCR đạt hiệu quả lý tưởng, thì cứ sau mỗi chu kỳ nhiệt cường độ huỳnh quang trong một ống phản ứng phải tăng gấp đôi, còn nếu ở hai ống phản ứng có số lượng bản DNA đích ban đầu cách nhau một hệ số pha loãng A, thì hai đường biểu diễn khuếch đại sẽ cách nhau n chu kỳ với cường độ huỳnh quang của hai ống phản ứng cũng sẽ cách nhau một hệ số pha loãng A = 2n. Như vậy, nếu độ pha loãng là 10 lần thì Ct của các độ pha loãng DNA đích của mẫu chuẩn liên tiếp nhau sẽ cách nhau là 3.32 chu kỳ (tính toán từ công thức trên với A = 10, sẽ tính được n = 3.32 chu kỳ). Trong biểu đồ chuẩn với các mẫu chuẩn cách nhau qua độ pha loãng hệ số 10 thì hiệu quả khuếch đại E được tính là bằng công thức 10-1/slope với slope chính là độ đốc của đường biểu diễn chuẩn. Một cách lý tưởng, hiệu quả khuếch đại E của PCR phải đạt được là 2 nghĩa là số lượng bản sao nhân bản từ bản DNA đích phải tăng gấp đôi sau mỗi chu kỳ nhiệt trong giai đoạn tăng trưởng lũy thừa, tức là 2 = 10-1/slope. Từ công thức này, chúng ta sẽ tính ra được đường biểu diễn chuẩn lý tưởng
phải có độ đốc (slope) là -3.32. Như vậy, chúng ta thấy độ dốc chính là sự cách biệt nhau về chu kỳ ngưỡng giữa các ống chuẩn có số lượng giảm dần theo hệ số pha loãng
- Hiệu quả khuếch đại cũng có thể tính bằng phần trăm (%) với công thức tính từ E là: E% = (E-1) x 100%. Với E đạt lý tưởng là 2 thì E% = 100 %. Đối với bất cứ một đường biểu diển chuẩn nào, đều có thể tính được hiệu quả khuếch đại E % = (10-1/slope
– 1) x 100 %. Do vậy nếu độ dốc của một đường biểu diễn chuẩn là -3.436 thì E sẽ là 10-(1/-3.436) = 1.954, vậy thì hiệu quả % của khuếch đại (hiệu quả PCR) sẽ là: E % = (1.954 – 1) x 100% = 95.4 %, có nghĩa là trong giai đoạn tăng trưởng lũy thừa số lượng bản sao sau mỗi chu kỳ nhiệt sẽ được nhân lên 1.954 lần hay hiệu quả PCR là 95.4 %. Dĩ nhiên hiệu quả PCR lý tưởng là 100 %, nhưng trong thực tế thì hiệu quả PCR chấp nhận được phải trong khoảng 90 % – 105 %. Người làm thí nghiệm phải biết dùng hiệu quả PCR hiển thị trong biểu đồ chuẩn để đánh giá thao tác pipetting để pha loãng mẫu của mình. Sai lầm đầu tiên có thể gặp trong thao tác pha loãng mẫu là mang lượng DNA đích từ mẫu nồng độ cao xuống mẫu có nồng độ thấp hơn, mà thường là do không thay đầu pipette sau khi mang thể tích từ nồng độ cao xuống nồng độ thấp, vì vậy mà số lượng bản DNA đích có trong các mẫu chuẩn được pha loãng sẽ nhiều hơn tính toán. Ví dụ nếu thao tác pha loãng chính xác, thì từ 10.000 copies xuống 1.000 copies rồi 100 copies sẽ chính xác như vậy; nhưng nếu không thay đầu pipette sau mỗi lần hút mẫu và lượng bản đích mang theo ở đầu pipette là 40% thì chúng ta sẽ có các số lượng sẽ là 10.000 copies xuống 4.000 copies rồi 1.600 copies. Vì vậy nên hiệu quả PCR được tính toán bằng giá trị E sẽ không đạt được lý tưởng 90 % đến 105 % mà sẽ cao hơn 105 %. Lý do là chu kỳ ngưỡng của các mẫu chuẩn sẽ bị gần nhau hơn làm cho độ dốc của đường biểu diễn chuẩn thấp đi. Ngược lại nếu thao tác pha loãng mà không cho đủ thể tích mẫu chuẩn hút từ mẫu có số lượng DNA chuẩn cao để pha mẫu chuẩn lượng thấp hơn (sai lầm này có thể gặp khi dùng các đầu pipette bị dính dung dịch trên thành nên không đẩy được hết lượng mẫu từ đầu pipette xuống mẫu kế), và chính như vậy nên chu kỳ ngưỡng của các mẫu chuẩn liên tiếp nhau sẽ cách xa nhau làm cho độ dốc của đường biểu diễn chuẩn thấp đi, và hiệu quả PCR sẽ dưới 95 %. Hiệu quả PCR cũng là một thông số rất có giá trị để nhà nghiên cứu tối ưu được kỹ thuật real-time PCR mà mình. Nếu hiệu quả PCR thấp thì nguyên do có thể là thiết kế mồi chưa đạt độ nhạy, do tách chiết DNA đích còn các ức chế ngăn cản phản ứng
khuếch đại. Nếu hiệu quả PCR cao thì nguyên do lại có thể là do sự bắt cặp không đặc hiệu của mồi hay của đoạn dò.
Như vậy, việc thiết lập biểu đồ chuẩn là rất cần thiết khi làm real-time PCR chẩn đoán, đặc biệt khi để xác định chính xác số lượng tác nhân đích ban đầu có trong mẫu thử. Biểu đồ chuẩn có công dụng trước tiên là đánh giá được thao tác pipetting của người làm thử nghiệm có đạt không. Đây là một việc là hết sức quan trọng vì nếu thao tác pipetting mà không chính xác thì sẽ không thể nào có được kết quả định lượng chính xác được. Do vậy sẽ thật là sai lầm và thiếu hiểu biết nếu nhà sản xuất bộ thuốc thử real-time PCR dành để định lượng tác nhân đích lại cung cấp sẵn các PCR mix có cho trước các lượng chuẩn của DNA đích, gọi là các bộ chuẩn, mà không để cho người làm thí nghiệm tự pha loãng các mẫu chuẩn để cho vào real-time PCR mix.
Sau khi đã đánh giá được thao tác pipetting của người làm thử nghiệm là đạt, người làm thử nghiệm sẽ sử dụng công dụng thứ hai của biểu đồ chuẩn, đó là xác định được số lượng tác nhân đích ban đầu có trong mẫu thử. Giá trị này được tính toán nhờ vào hàm số biểu thị mối tương quan giữa chu kỳ ngưỡng (Y = Ct) với log10 của số lượng bản DNA đích ban đầu có trong ống phản ứng (X = log10 Sq). Hàm số đó là: Y = [slope (X)] + intercept, và các thông số slope và intercept đều hiển thị trên biểu đồ chuẩn. Từ hàm số này, người làm thí nghiệm sẽ hiểu được tại sao máy tính hiển thị được Sq, đó là nhờ tính toán từ Sq = 10[(Ct – intercept)/slope]. Từ Sq này, người làm thí nghiệm sẽ tính được số lượng tác nhân đích ban đầu có trong mẫu thử dựa vào hiệu quả tách chiết nucleic acid đích từ mẫu thử và các pha loãng nếu có trong quá trình tách chiết củng như thực hiện real-time PCR. Ví dụ: khi sử dụng các bộ thuốc thử phát hiện và định lượng HCV do công ty Nam Khoa sản xuất thì số copies HCV có trong 1 ml huyết thanh thử nghiệm sẽ bằng N x 71 với N là số copies định lượng được trong ống phản ứng, hay khi khi sử dụng các bộ thuốc thử phát hiện và định lượng HBV do công ty Nam Khoa sản xuất thì số copies HBV có trong 1 ml huyết thanh thử nghiệm sẽ bằng N x 50 với N là số copies định lượng được trong ống phản ứng (xem phụ lục để biết cách tính toán để có các hệ số này).
Máy real-time PCR
Điều kiện đầu tiên để có thể thực hiện được real-time PCR là phải có máy real-time PCR. Máy này cũng có một buồng ủ nhiệt chạy được chương trình luân nhiệt như máy PCR bình thường, nhưng có thêm một thiết bị được gọi là thiết bị real-time. Đây là một thiết bị quang học có hai chức năng: (1) Có các nguồn sáng phát ra được các tia sáng kích thích (excitation light) có bước sóng xác định lên các tube phản ứng real-time PCR;
- Có được camera hay cảm biến quang ghi nhận được ánh sáng huỳnh quang phát ra (emission light) từ các tube phản ứng real-time PCR khi các tube phản ứng này được chiếu các tia sáng kích thích.
Tùy theo hãng và model sản xuất mà có nhiều kiểu thiết bị real-time khác nhau. Các kiểu thiết bị này khác nhau về cách phát ra nguồn sáng kích thích và cách ghi nhận được ánh sáng huỳnh quang được phát ra từ các tube phản ứng. Tóm tắt có các kiểu sau đây:
1. Sử dụng nguồn sáng kích thích là đèn tungstene và dùng các kính lọc để chiếu nguồn sáng có độ dài sóng nhất định lên toàn bộ các giếng chứa tube phản ứng
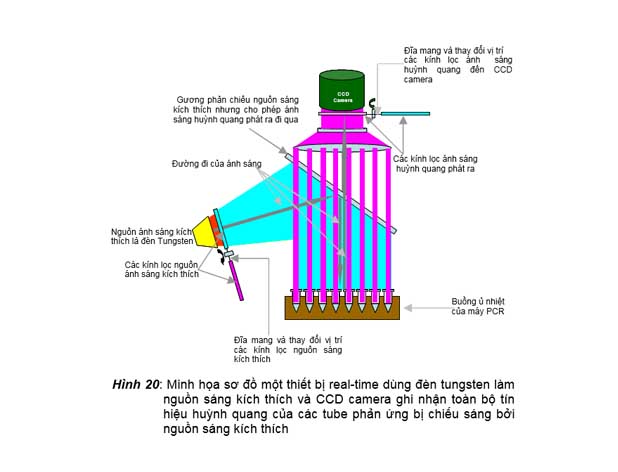
một thiết bị real-time dùng đèn tungstene làm nguồn sáng kích thích. Nguồn sáng này đi qua một kính lọc màu được người sử dụng chọn (bằng chương trình điều khiển để xoay đĩa chọn kính lọc màu thích hợp đến đúng vị trí) chỉ
cho một loại ánh sáng có độ
Gương phản chiếu nguồn sáng kích thích nhưng cho phép ánh sáng huỳnh quang phát ra đi qua
Đường đi của ánh sáng
Nguồn ánh sáng kích thích là đèn Tungsten
Các kính lọc nguồn ánh sáng kích thích
CCD
Camera
Đĩa mang và thay đổi vị trí
các kính lọc ánh sáng huỳnh quang đến CCD camera
Các kính lọc ánh sáng huỳnh quang phát ra
Buồng ủ nhiệt của máy PCR
dài sóng thích hợp đi qua. Ánh sáng kích thích có độ dài sóng chọn lọc này được gương dichroic phản chiếu
Đĩa mang và thay đổi vị trí các kính lọc nguồn sáng kích thích
Hình 20: Minh họa sơ đồ một thiết bị real-time dùng đèn tungsten làm nguồn sáng kích thích và CCD camera ghi nhận toàn bộ tín hiệu huỳnh quang của các tube phản ứng bị chiếu sáng bởi nguồn sáng kích thích
xuống buồng ủ nhiệt có các tube phản ứng. Trong tube phản ứng có chứa một loại hóa chất có khả năng phát huỳnh quang, nếu có sự hiện diện của sợi đôi DNA được khuếch
đại từ DNA đích và bị chiếu sáng bởi ánh sáng kích thích có độ dài sóng thích hợp. Ánh sáng huỳnh quang phát ra từ tube phản ứng sẽ đi qua gương dichroic, qua kính lọc màu huỳnh quang để được ghi nhận bởi một CCD camrea. CCD camera này sẽ chụp hình nguyên buồng ủ nhiệt của máy PCR để ghi nhận tín hiệu và cường độ huỳnh quang được phát ra từ các tube phản ứng qua mỗi chu kỳ nhiệt và đưa về bộ vi xử lý của máy vi tính rồi hiển thị real-time lên màn hình bằng biểu đồ để người làm thí nghiệm thấy được cường độ của tín hiệu huỳnh quang phát ra từ mỗi giếng phản ứng qua các chu kỳ nhiệt. Thiết bị real-time kiểu này có thể thấy được trong các máy real- time PCR thế hệ IQ (ví dụ IQ, MyIQ, IQ5) của hãng Biorad.
2. Thiết bị real-time dùng sợi quang học (fiber optic cables) để đưa nguồn sáng kích thích đến các tube phản ứng
Trong thiết bị này, nguồn sáng kích thích có thể là đèn laser, hay đèn tungstene, được các sợi quang học dẫn trực tiếp đến các tube phản ứng, và các sợi quang học học đồng thời
cũng làm luôn nhiệm vụ dẫn ánh sáng huỳnh quang phát ra đến CCD camera.
Nguồn sáng laser hay tungstene
Buồng ủ nhiệt
Fiber optic cable
Fiber optic cable
Thấu kính nhỏ Tube phản ứng
Buồng ủ nhiệt
Thiết bị real- time kiểu
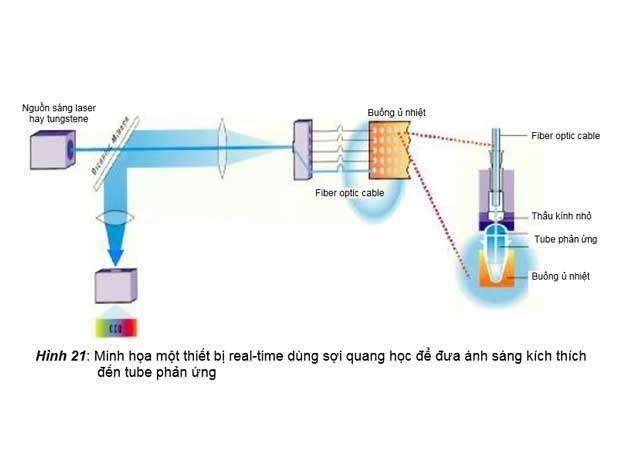
này có thể được thấy trong các máy realtime PCR của hãng ABI, như các máy real- time ABI 7000 hay ABI 7.500 (hình 21).
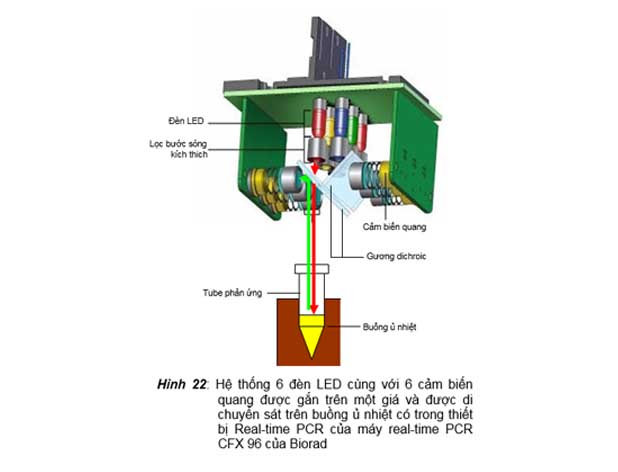
3. Thiết bị real-time dùng đèn led làm nguồn sáng kích thích
Trong thiết bị này, nguồn sáng kích thích được sử dụng là các đèn LED được gắn trên một giá và giá này di chuyển sát trên buồng ủ nhiệt (hình 22) để chiếu ánh sánh kích thích lên các tube phản ứng và nhận ánh sáng huỳnh quang phát ra (máy real-time PCR model CFX 96 của Biorad), hay là được cố định tại một vị trí trong buồng ủ nhiệt và các tube phản ứng được di chuyển đến vị trí này nhờ các tube phản ứng được gắn
trên một giá xoay tròn (máy luân nhiệt Ligh-cycler của Roche hay Rotor Gene của Corbett Research). Lợi điểm của thiết bị real-time này là đời sống của đèn LED có thể kéo dài rất lâu, có thể đến hàng chục ngàn giờ.
Hóa chất và thuốc thử cho real-time PCR
Chìa khóa kỹ thuật chính của hóa chất và thuốc thử có trong tube phản ứng real-time PCR chính là chất huỳnh quang được thêm
vào PCR mix. Chất huỳnh quang này phải
Đèn LED Lọc bước sóng
kích thich
Tube phản ứng
Cảm biến quang Gương dichroic
Buồng ủ nhiệt
làm thế nào để tube phản ứng có thể phát được huỳnh quang khi bị chiếu bởi nguồn sáng kích thích một khi trong tube phản ứng
này có sự hiện diện sản phẩm khuếch đại của PCR từ DNA đích, và sẽ không thể phát được huỳnh quang nếu không có sản phẩm khuếch đại trong ống. Cho đến nay, các nhà khoa học đã tìm ra được nhiều chất huỳnh quang như vậy. Trong phạm vi nội dung của bài này, chúng tôi chỉ xin trình bày một số chất huỳnh quang thường được sử dụng nhất.
1. Chất phát huỳnh quang là một loại màu huỳnh quang chèn vào sợi đôi DNA
Màu huỳnh quang chèn vào sợi đôi DNA là một loại màu huỳnh quang có ái lực rất cao khi có sự hiện diện của sợi đôi DNA và ái lực này là do khả năng chèn của màu vào giữa sợi đôi DNA và làm cho sợi đôi DNA phát được ánh sáng huỳnh quang khi nhận được nguồn sáng kích thích.
Nguyên tắc kỹ thuật real-time PCR dùng màu huỳnh quang chèn
Nguyên tắc của kỹ thuật này là khi không có sự hiện diện sản phẩm khuếch đại của PCR, chất huỳnh quang bị phân tán trong dung dịch PCR mix, do vậy mà tube phản ứng sẽ không bị phát huỳnh quang hay chỉ phát huỳnh quang không đáng kể khi bị chiếu bởi nguồn sáng kích thích. Nhưng khi có sự hiện diện sản phẩm khuếch đại của PCR thì màu huỳnh quang sẽ bị chèn vào và tập trung trên các sợi đôi DNA của sản phẩm khuếch đại và sẽ làm cho tube phản ứng bị phát huỳnh quang khi bị chiếu bởi
nguồn sáng kích thích. Giống như trên phi cơ nhìn xuống mặt biển vào ban đêm mà trên mặt biển có rải rác các thuyền con sáng đèn. Nếu các thuyền con này cách xa nhau thì chúng ta sẽ thấy mặt biển tối đen, nhưng nếu chúng tập trung lại với nhau thì chúng ta sẽ thấy trên mặt biển có những vùng sáng do tập trung ánh sáng từ các thuyền.
Khởi thủy ethidium bromide được sử dụng cho nguyên tắc real-time PCR này, nhưng sau này các nhà khoa học sử dụng SYBR I vì các ưu điểm vượt trội hơn như màu huỳnh quang nền rất thấp, khả năng chèn vào sợi đôi cao nhưng không làm cho sợi đôi bị gắn chặt vào nhau khi bị biến tính nhờ vậy mà ảnh hưởng ít lên hiệu quả
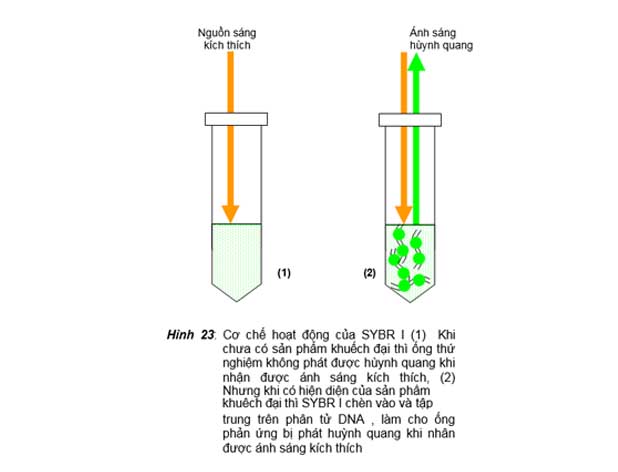
PCR. Hình 23 minh họa nguyên tắc hoạt động của real-time PCR dùng màu chèn là SYBR I làm chất phát huỳnh quang. Phổ quang của SYBR là: l = 488 cho nguồn sáng kích thích và l = 522 cho huỳnh quang phát ra.
Thiết kế mồi cho real-time PCR dùng màu huỳnh quang chèn
Thiết kế mồi cho real-time PCR dùng
màu huỳnh quang chèn cũng theo các qui luật thiết kế mồi chung cho PCR, đó là: (1) tránh hiện tượng chân tóc ở đầu 5’ hay 3’;
(2) tránh bắt cặp giữa primer với nhau hay cùng primer với nhau, nhất là ngay ở đầu 3’; (3) Tránh bắt cặp không đặc hiệu trên trình tự đích; (4) thành phần GC trong khoảng 40-60%; (5) Trong trình tự mồi, tránh lặp liên tiếp trên 3 base G hay C; (6)
Nguồn sáng kích thích
(1) (2)
Ánh sáng hùynh quang
Nên thiết kế để đầu 3’ có base là G hay C
để tránh sản phẩm khuếch đại không đặc hiệu; (7) Nên thiết kế để nhiệt độ bắt cặp
Hình 23: Cơ chế hoạt động của SYBR I (1) Khi chưa có sản phẩm khuếch đại thì ống thứ nghiệm không phát được hùynh quang khi nhận được ánh sáng kích thích, (2) Nhưng khi có hiện diện của sản phẩm
o o khuếch đại thì SYBR I chèn vào và tập
tối hảo là từ 55 C đến 65 C để không bị khuếch đại không đặc hiệu vì nhiệt độ bắt cặp quá thấp. Ngoài ra, đối với real-time
trung trên phân tử DNA , làm cho ống phản ứng bị phát huỳnh quang khi nhân được ánh sáng kích thích
PCR thì lưu ý thêm một yếu tố kỹ thuật nữa, đó là sản phẩm khuếch đại: nên thiết kế mồi để có sản phẩm khuếch đại từ 75 đến 200 bps. Không nên ít hơn 75 bps vì như vậy sẽ khó phân biệt được sản phẩm khuếch đại đặc hiệu với sản phẩm khuếch đại của
dimer-primer (xem phần phân tích melt curve), không nên dài quá 200 bps vì như vậy sẽ khó đạt được hiệu quả PCR lý tưởng và như vậy thì biểu đồ chuẩn sẽ không đạt để có thể cho được kết quả định lượng chính xác (xem phần biểu đồ chuẩn của real-time PCR).
Cách pha real-time PCR mix sử dụng màu huỳnh quang chèn là SYBR I
Để thực hiện được kỹ thuật real-time PCR sử dụng SYBR I làm chất phát huỳnh quang thì người làm thí nghiệm phải pha PCR mix trong đó có thêm SYBR I với nồng độ 1X (pha từ stock 10.000X), nồng độ MgCl2 đến 3mM, và thêm một ít DMSO, BSA… Ngoài ra, tùy thuộc vào thiết bị real-time PCR có đòi hỏi PCR mix phải thêm màu huỳnh quanh nền hay không để thiết bị nhận diện được vị trí và xác định được giá trị ngưỡng huỳnh quang ban đầu của từng giếng thử nghiệm, mà người làm thí nghiệm có cho thêm chất huỳnh quang nền vào PCR mix hay không. Ví dụ với các máy real- time thế hệ IQ của Biorad thì huỳnh quang nền cho vào PCR mix là fluorescent, còn với các máy ABI 7000 thì huỳnh quang nền là ROX. Để có thể thực hiện pha real-time PCR mix một cách đơn giản mà lại hoạt động một cách hiệu quả, người làm thí nghiệm có thể pha từ real-time PCR master mix do các hãng sản xuất có uy tín cung cấp có chứa sẵn SYBR I, enzyme Taq polymerase, dNTP và MgCl2. Với PCR master mix gọi là SYBR PCR master mix này, người làm thí nghiệm chỉ cho thêm mồi, nồng độ 10-25 pm cho một thể tích phản ứng, và nếu cần thì cho thêm dUTP và UNG để chống ngoại nhiễm, là có được real-time PCR mix để thực hiện real-time PCR.
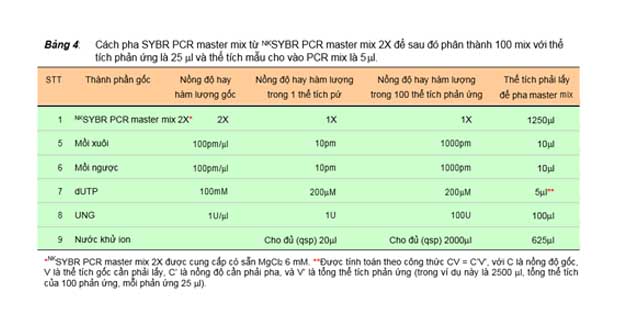
Bảng 4: Cách pha SYBR PCR master mix từ NKSYBR PCR master mix 2X để sau đó phân thành 100 mix với thể tích phản ứng là 25 ml và thể tích mẫu cho vào PCR mix là 5 ml.
STT | Thành phần gốc | Nồng độ hay hàm lượng gốc | Nồng độ hay hàm lượng trong 1 thể tích pứ | Nồng độ hay hàm lượng trong 100 thể tích phản ứng | Thể tích phải để pha master | lấy mix | |
1 | NKSYBR PCR | master | mix 2X* 2X | 1X | 1X | 1250ml | |
5 | Mồi xuôi | 100pm/ml | 10pm | 1000pm | 10ml | ||
6 | Mồi ngược | 100pm/ml | 10pm | 1000pm | 10ml | ||
7 | dUTP | 100mM | 200mM | 200mM | 5ml** | ||
8 | UNG | 1U/ml | 1U | 100U | 100ml | ||
9 | Nước khử ion | Cho đủ (qsp) 20ml | Cho đủ (qsp) 2000ml | 625ml | |||
*NKSYBR PCR master mix 2X được cung cấp có sẵn MgCl2 6 mM. **Được tính toán theo công thức CV = C’V’, với C là nồng độ gốc, V là thể tích gốc cần phải lấy, C’ là nồng độ cần phải pha, và V’ là tổng thể tích phản ứng (trong ví dụ này là 2500 ml, tổng thể tích của 100 phản ứng, mỗi phản ứng 25 ml).
trình bày cách pha một SYBR PCR master mix cho 100 mix từ NKSYBR PCR master mix 2X do công ty Nam Khoa sản xuất.
Chương trình luân nhiệt real-time PCR dùng SYBR I là màu chèn
Với real-time PCR sử dụng SYBR I làm chất phát huỳnh quang; thiết bị real-time sẽ ghi nhận được sự phát huỳnh quang tối đa từ ống phản ứng, khi nhận ánh sáng kích thích, là ở cuối giai đoạn nhiệt độ kéo dài của chu kỳ PCR. Do vậy một chương trình luân nhiệt cho real-time PCR sử dụng SYBR có thể là: 1 chu kỳ: 40oC/10’ (nếu trong PCR mix có dUTP và UNG), 1 chu kỳ: 95oC/5’ (nếu dùng hot-start Taq polymerase1); 35-40 chu kỳ, là chu kỳ luân nhiệt cho PCR gồm 3 giai đoạn nhiệt độ: 94oC/15-30”, 55oC – 65oC/30-60”, 72oC/30-60”. Trong các chu kỳ luân nhiệt PCR này, chọn lệnh để thiết bị real-time phát ra nguồn sáng kích thích và ghi nhận ánh sáng huỳnh quang phát ra từ ống phản ứng trong giai đoạn nhiệt độ bắt cặp 55oC – 65oC, máy sẽ tự động thực hiện hai chức năng quang học quan trọng này ở giai đoạn cuối của bước nhiệt độ bắt cặp.
Chương trình phân tích nhiệt độ chảy
Vì SYBR I là màu chèn cho bất cứ sợi đôi DNA nào xuất hiện trong ống phản ứng sau các chu kỳ nhiệt kể cả các sợi đôi DNA không
phải khuếch đại đặc hiệu từ DNA đích, do vậy sự xuất hiện đường biểu diễn khuếch đại trong ống phản ứng sẽ không đặc hiệu 100 % cho sự có mặt của sản phẩm khuếch đại đặc hiệu từ DNA đích. Như vậy thì trong ống phản ứng, sản phẩm khuếch đại nào thường có thể xuất hiện mà không phải là sản phẩm khuếch đại từ DNA đích? Đó chính là các sản phẩm khuếch đại từ
3’
5’
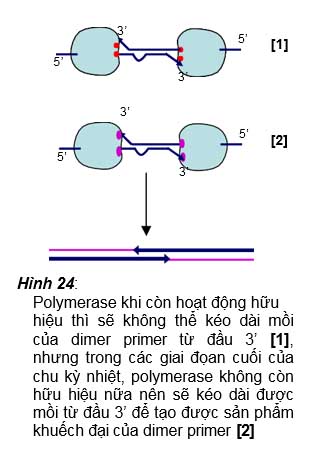
các dimer primer, thường xảy ra vào các giai đoạn cuối của các chu kỳ nhiệt, khi mà enzyme Taq polymerase hoạt động không còn hiệu quả và đặc hiệu nữa. Dimer primer là sự bắt cặp lẫn nhau giữa mồi do
Polymerase khi còn hoạt động hữu
hiệu thì sẽ không thể kéo dài mồi của dimer primer từ đầu 3’ [1], nhưng trong các giai đọan cuối của chu kỳ nhiệt, polymerase không còn hữu hiệu nữa nên sẽ kéo dài được mồi từ đầu 3’ để tạo được sản phẩm khuếch đại của dimer primer [2]
1Hot start Taq polymerase là Taq polymerase mà ở nhiệt độ thường bị bất hoạt vì có kháng thể đặc hiệu (hay hóa chất) bám lên điểm hoạt động của enzyme. Khi bị đun lên 95oC trong 5 phút thì kháng thể dặc hiệu sẽ bị phát hủy, Enzyme sẽ hoạt động. Hot start Taq polymerase thường được sử dụng để tránh các bắt cặp không đặc hiệu ở nhiệt độ dưới nhiệt độ bắt cặp đặc hiệu của mồi
có những vị trí trên trình tự mồi mang những base bổ sung được với nhau. Tuy nhiên trong các chu kỳ đầu khi mà enzyme polymerase còn hoạt động hiệu quả thì sẽ không tạo được sản phẩm khuếch đại đặc hiệu do sự kéo dài của hai mồi khi chúng bắt cặp lẫn nhau chỉ vài vị trí nucleotide mà không bắt cặp được ở đầu 3’ (hình 24 [1]). Tuy nhiên trong các giai đoạn cuối của PCR, enzyme polymerase đã trở nên hoạt động kém hữu hiệu nên nó vẫn có thể kéo dài được các mồi khi chúng bắt cặp lẫn nhau dù ở đầu 3’ của mồi vẫn không có bắt cặp đặt hiệu với nhau, chính vì vậy nên mới tạo được các sản phẩm khuếch đại không đặc hiệu từ các dimer primer (hình 24 [2]).
Sản phẩm khuếch đại từ dimer primer sẽ khác biệt với sản phẩm khuếch đại từ DNA đích chính là ở chiều dài, nếu từ dimer primer thì chiều dài sẽ không bao giờ vượt quá 50 – 55 bps, còn nếu từ DNA đích thì chiều dài thường quá 100 bps. Như vậy, để có thể phân biệt được được đường biểu diễn khuếch đại của ống phản ứng là của sản phẩm khuếch đại đặc hiệu từ bản DNA đích hay là từ dimer primer,
phải dựa vào một tính chất đặc trưng giúp phân biệt được chiều dài của hai loại sản phẩm khuếch đại này, và tính chất đặc trưng đó chính là nhiệt độ chảy (melting To, Tm), là nhiệt độ làm cho sợi đôi DNA bị biến tính 50 % tức là có 50 % số sợi đôi bị biến tính hoàn toàn. Nhiệt độ chảy của sợi đôi DNA tùy thuộc vào thành phần GC và chiều dài của nó: Thành phần GC càng cao thì Tm càng cao, sợi đôi càng dài thì Tm càng cao. Sản phẩm khuếch đại từ DNA đích dài hơn nên sẽ có Tm cao hơn là Tm của sản phẩm khuếch đại từ dimer primer. Để biết được Tm của sản phẩm khuếch đại có trong ống phản ứng, sau khi hoàn tất các chu kỳ luân nhiệt của real-time PCR, người làm thí nghiệm sẽ tiếp tục
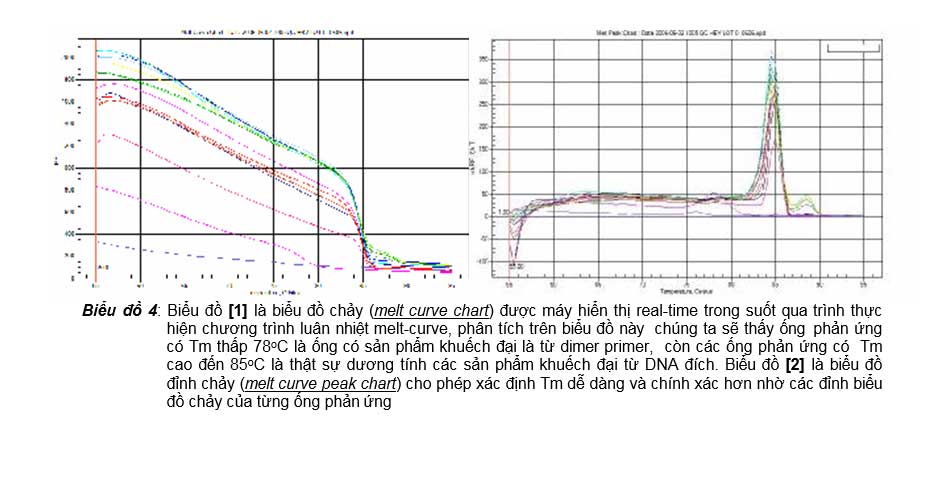
Biểu đồ 4: Biểu đồ [1] là biểu đồ chảy (melt curve chart) được máy hiển thị real-time trong suốt qua trình thực hiện chương trình luân nhiệt melt-curve, phân tích trên biểu đồ này chúng ta sẽ thấy ống phản ứng có Tm thấp 78oC là ống có sản phẩm khuếch đại là từ dimer primer, còn các ống phản ứng có Tm cao đến 85oC là thật sự dương tính các sản phẩm khuếch đại từ DNA đích. Biểu đồ [2] là biểu đồ đỉnh chảy (melt curve peak chart) cho phép xác định Tm dễ dàng và chính xác hơn nhờ các đỉnh biểu đồ chảy của từng ống phản ứng
cho máy chạy thêm một chương trình phân tích nhiệt độ chảy, gọi là chương trình melt-curve. Tùy thuộc vào từng loại máy real-time PCR mà người làm thí nghiệm sử dụng chương trình melt-curve được cài sẵn trong máy, hay là tự tạo chương trình melt curve mà mình thấy thích hợp hơn. Đối với các máy real-time PCR thế hệ iQ của Biorad thì chương trình melt-curve là, trước hết buồng ủ nhiệt sẽ đưa nhiệt độ lên 95oC trong 1 phút để làm biến tính hoàn toàn các sản phẩm khuếch đại có trong ống phản ứng, rồi hạ xuống nhiệt độ 55oC trong 1 phút để tất cả các sản phẩm khuếch đại này bắt cặp hoàn toàn thành sợi đôi. Sau đó thực hiện chương trình melt-curve đưa nhiệt độ buồng ủ nhiệt từ 55oC lên 95oC trong 80 bước tăng nhiệt độ, mỗi bước tăng 0.5oC và giữ trong 10 giây; đồng thời trong mỗi bước này, thiết bị real-time cũng sẽ chiếu nguồn sáng kích thích và ghi nhận và hiển thị trên đồ thị cường độ ánh sáng huỳnh quang phát ra từ các ống phản ứng lên một biểu đồ chảy (melt curve chart). Phân tích trên biểu đồ hiển thị real-time này, chúng ta sẽ thấy đối với từng ống phản ứng thì cường độ huỳnh quang phát ra sẽ giảm dần theo các bước tăng nhiệt độ trong buồng ủ nhiệt, lý do là các sản phẩm khuếch đại có trong ống phản ứng sẽ bị biến tính dần dần theo sự gia tăng nhiệt độ trong buồng ủ nhiệt. Đến một bước tăng nhiệt độ mà ở đó trùng khớp với Tm của sản phẩm khuếch đại trong ống phản ứng thì cường độ huỳnh quang sẽ giảm đột ngột vì có đến 50 % sản phẩm khuếch đại này bị biến tính thành sợi đơn cùng một lúc, do vậy ở bước nhiệt độ này chúng ta sẽ thấy đường biểu diễn cường độ huỳnh quang phát ra từ ống phản ứng không còn là một đường thẳng đi xuống đều như trước mà bị chúi xuống thấp hơn nhiều so với độ dốc của nó. Nhờ vậy mà người làm thí nghiệm khi quan sát diễn tiến của quá trình vẽ real-time biểu đồ chảy, sẽ hoàn toàn có thể có thể xác định được Tm của sản phẩm khuếch đại có trong ống phản ứng (biểu đồ 4 [1]). Để giúp xác định được dễ dàng hơn Tm, sau khi hoàn tất chương trình melt-curve, máy real-time PCR sẽ thay đổi hiển thị biểu đồ chảy thành một đồ khác gọi là biểu đồ đỉnh chảy (melt curve peak, biểu đồ 4 [2]), trong biểu đồ này trục hoành (X) vẫn là biến thiên nhiệt độ của buồng ủ, nhưng trục tung (Y) thì thay biến thiên cường độ huỳnh quang (RFU) thành biến thiên tỷ lệ giảm cường độ huỳnh quang so với tăng nhiệt độ của buồng ủ nhiệt (DRFU/DT). Nhờ sự thay đổi này, chúng ta sẽ thấy trị số của
DRFU/DT hầu như không biến đổi khi sự gia tăng nhiệt độ của buồng ủ nhiệt chưa đạt đến Tm, nhưng khi nhiệt độ buồng ủ nhiệt đạt đến Tm thì sẽ có sự gia tăng đáng kể trị số DRFU/DT, và trên biểu đồ chúng ta sẽ thấy một đỉnh (peak) của trị số này. Giá trị
nhiệt độ buổng ủ tương ứng với đỉnh của đường biểu diễn này cho ta được trị số Tm của sản phẩm khuếch đại có trong ông phản ứng. Như vậy với phân tích melt curve trong phương pháp real-time PCR sử dụng SYBR Green I, người làm thí nghiệm có thể phân biệt được sản phẩm khuếch đại có trong ống phản ứng là từ DNA đích hay là từ dimer primer, do đó mà đã khắc phục được nhược điểm không đặc hiệu của chất phát huỳnh quang này trong phát hiện sản phẩm khuếch đại xuất hiện trong ống phản ứng trong quá trình luân nhiệt.
Kỹ thuật phân tích phân giải cao nhiệt độ chảy (High resolution melting, HRM)
Mặc dù có ưu điểm hơn ethidium bromide, nhưng SYBR green I cũng còn một nhược điểm rất quan trọng, đó là tín hiệu huỳnh quang phát ra không cao và lại có thể ức chế PCR. Chính vì nhược điểm này mà đường biểu diễn đỉnh chảy sẽ không cao, độ phân giải các đỉnh chảy của các sản phẩm khuếch đại có trình tự khác biệt hay/và chiều dài khác biệt cũng rất thấp, không thể phân biệt được chúng với nhau. Chính vì vậy mà real-time PCR sử dụng SYBR I làm chất phát huỳnh quang không thể ứng dụng trong multiplex real-time PCR, trong real-time PCR phát hiện các SNP (single nucleotide polymorphism), hay real-time PCR định được genotype.
Hiện nay đã có những tiến bộ đáng kể trong phát hiện các màu huỳnh quang chèn khắc phục được các nhược điểm của SYBR green I. Các màu này có khả năng phát huỳnh quang rất cao và hơn nữa lại hoàn toàn không ức chế PCR nên độ phân giải của các biểu đồ đỉnh chảy rất cao, do vậy được gọi là các màu HRM (high resolution melting dye). Bảng 5 liệt kê một số chất huỳnh quang HRM hiện nay đang được các nhà nghiên cứu ứng dụng, và sự lựa chọn màu nào là tùy thuộc vào kênh ánh sáng kích thích và ánh sáng huỳnh quang mà máy real-time PCR họ đang sử dụng có thể thực hiện được trong quá trình chạy luân nhiệt. Có thể tóm tắt các lựa chọn này như sau: SYTO9 hay LCGreen thì chỉ dành cho máy real-time PCR Rotor Gene của Corbett hay Light Cycler của Roche vì các máy này mới có kênh màu thích hợp; EVAGreen hay BEBO thì có thể thích hợp trên các máy real-time PCR thông dụng và rất tối ưu trên real-time PCR của Biorad hay Applied Biosystem (dùng đúng kênh màu FAM hay SYBR green I); BOXTO thì dùng kênh màu Joe có trên nhiều máy real-time PCR thông dụng, có thể kết hợp với real-time PCR dùng Beacon probe gắn chất phát huỳnh
quang là FAM, nhờ vậy mà có thể định lượng DNA đích bằng FAM và phân tích HRM curve với kênh Joe.
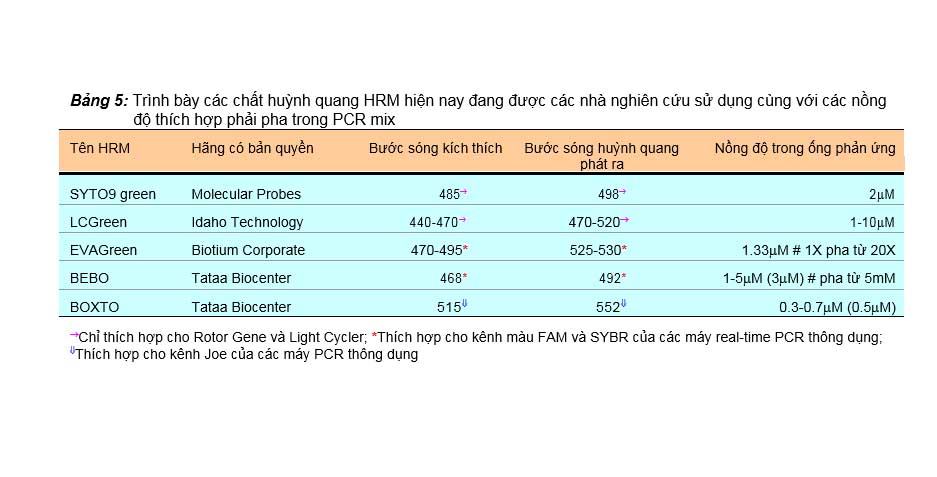
Bảng 5: Trình bày các chất huỳnh quang HRM hiện nay đang được các nhà nghiên cứu sử dụng cùng với các nồng
độ thích hợp phải pha trong PCR mix
Tên HRM | Hãng có bản quyền | Bước | sóng kích | thích | Bước | sóng huỳnh phát ra | quang | Nồng độ trong ống phản ứng |
SYTO9 green | Molecular Probes | 485® | 498® | 2mM | ||||
LCGreen | Idaho Technology | 440-470® | 470-520® | 1-10mM | ||||
EVAGreen | Biotium Corporate | 470-495* | 525-530* | 1.33mM # 1X pha từ 20X | ||||
BEBO | Tataa Biocenter | 468* | 492* | 1-5mM (3mM) # pha từ 5mM | ||||
BOXTO | Tataa Biocenter | 515ß | 552ß | 0.3-0.7mM (0.5mM) | ||||
®Chỉ thích hợp cho Rotor Gene và Light Cycler; *Thích hợp cho kênh màu FAM và SYBR của các máy real-time PCR thông dụng;
ßThích hợp cho kênh Joe của các máy PCR thông dụng
Với real-time PCR dùng HRM làm màu chèn, nhà nghiên cứu cũng như người làm thử nghiệm hoàn toàn có thể thực hiện real-time PCR để không chỉ định lượng một cách đặc hiệu được tác nhân đích có trong mẫu thử, mà còn có thể thực hiện được multiplex real-time PCR để phát hiện nhiều tác nhân đích, phát hiện các đột biến – thậm chí SNP với chỉ 1 nucleotide khác biệt, phát hiện và xác định được genotype. Chìa khóa
chính của các bước tiến này là khả năng
phân biệt được sự khác biệt không chỉ về chiều dài mà cả sự khác biệt trình tự có khi đến 1 nucleotide của các sản phẩm khuếch đại có trong ống phản ứng nhờ bản chất ưu việt của các màu HRM là không ức chế PCR và đồng thời cường độ phát phát
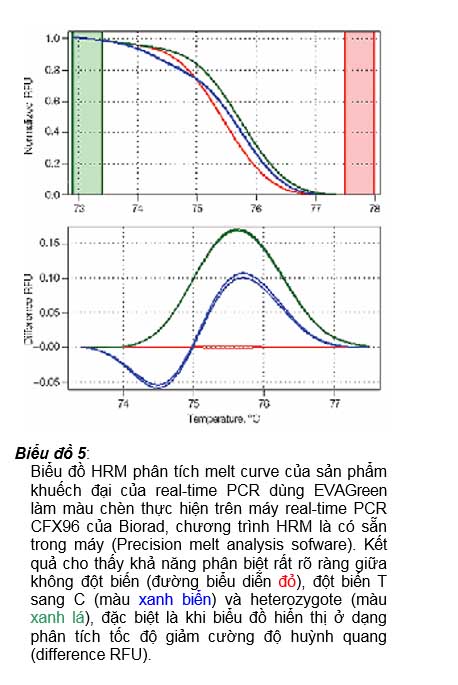
Biểu đồ 5:
Biểu đồ HRM phân tích melt curve của sản phẩm khuếch đại của real-time PCR dùng EVAGreen làm màu chèn thực hiện trên máy real-time PCR CFX96 của Biorad, chương trình HRM là có sẵn trong máy (Precision melt analysis sofware). Kết quả cho thấy khả năng phân biệt rất rõ ràng giữa không đột biến (đường biểu diễn đỏ), đột biến T sang C (màu xanh biển) và heterozygote (màu xanh lá), đặc biệt là khi biểu đồ hiển thị ở dạng phân tích tốc độ giảm cường độ huỳnh quang (difference RFU).
huỳnh quang của một phân tử màu HRM khi chèn vào sợi đôi DNA có thể tăng đến trên 250 lần so với khi tự do trong dung dịch. Chính nhờ vậy mà người làm thí nghiệm khi chạy chương trình luân nhiệt phân tích melt curve độ phân giải cao (gọi là chương trình HRM) – tức là chỉ chạy luân nhiệt phân tích melt curve trong một khoảng cách biệt nhiệt độ hẹp (ví dụ 73oC đến 78oC là khoảng nhiệt độ mà sản phẩm khuếch đạt đạt
Tm, thay vì 55oC đến 95oC như trong chương trình luân nhiệt phân tích melve curve bình thường để dò Tm) – kết quả biểu đồ đỉnh chảy hoàn toàn đủ phân giải cao để phân biệt sự khác biệt của các sản phẩm khuếch đại có trong ống phản ứng. Biểu đồ 5 minh họa một kết quả cho thấy có thể phân biệt được sự khác biệt chỉ 1 nucleotide, kể cả tình trạng heterozygote của các sản phẩm khuếch đại qua biểu đồ HRM.
2. Real-time PCR sử dụng probe làm chất phát huỳnh quang
Probe được dịch là “đoạn dò” hay “dò”, đó là những đoạn oligonucletides sợi đơn có trình tự có thể bắt cặp bổ sung với một trình tự đặc hiệu trên DNA đích (trong PCR, đó là sản phẩm khuếch đại đặc hiệu từ DNA đích). Sử dụng probe làm chất phát huỳnh quang dựa trên nguyên tắc là khi có mặt sản phẩm khuếch đại đặc hiệu trong ống phản ứng thì sẽ có sự bắt cặp của probe lên trình tự đặc hiệu của sản phẩm khuếch đại, và khi có sự bắt cặp này thì sẽ có sự phát huỳnh quang từ ống phản ứng khi nó nhận được nguồn sáng kích thích. Có nhiều loại probe, và thậm chí cả primers được sử dụng làm chất phát huỳnh quang cho real-time PCR. Trong phạm vi bài này, chúng tôi xin trình bày kỹ một số thường được sử dụng hiện nay.
- Real-time PCR sử dụng Taqman probe
Nguyên tắc kỹ thuật real-time PCR sử dụng Taqman probe
Trong kỹ thuật này, real-time PCR mix ngoài các thành phần cơ bản của PCR mix, còn chứa hai thành phần quan trọng để probe có thể phát huỳnh quang được khi có sự hiện diện của sản phẩm khuếch đại đặc hiệu từ DNA đích, đó là: (1) Taqman probe, là những oligonucleotides có trình tự bổ sung với một trình tự đặc hiệu trên DNA đích, và trình tự này dài khoảng 24 đến 30 bases với đầu 5’ có gắn chất phát huỳnh quang (gọi là reporter) còn đầu 3’ có gắn chất hấp phụ tương ứng (gọi là quencher) để hấp phụ được ánh sáng huỳnh quang được phát ra từ reporter. (2) Enzyme Taq polymerase có hoạt tính 5’-3’ exonuclease để có khả năng thủy giải cắt bỏ probe khi probe này bắt cặp lên sợi khuôn và cản đầu 3’ của mồi khi enzyme kéo dài mồi tổng hợp sợi bổ sung. Cơ chế phát huỳnh quang của Taqman probe trong các chu kỳ nhiệt được trình bày minh họa trong hình 25. Có thể tóm tắt như sau: (1) Khi chưa có sự xuất hiện của sản phẩm khuếch đại đặc hiệu từ DNA đích thì Taqman probe vẫn còn nguyên vẹn do vậy mà huỳnh quang phát ra được từ reporter ở đầu 5’ sẽ bị quencher ở đầu 3’ của probe hấp
phụ, ống thử nghiệm sẽ không phát được huỳnh quang khi nhận được nguồn sáng kích thích. (2) Khi bắt đầu có sự xuất hiện sản phẩm khuếch đại đặc hiệu thì Taqman probe sẽ bắt cặp vào sợi khuôn của sản phẩm này ở giai đoạn nhiệt độ bắt cặp và sẽ bị enzyme Taq polymerase cắt bỏ để kéo dài mồi tổng hợp nên sợi bổ sung, do vậy mà reporter của Taqman probe sẽ bị cắt rời xa quencher và phát được huỳnh quang khi nhận được nguồn sáng kích thích. Càng nhiều sản phẩm khuếch đại xuất hiện thì số lượng reporter tự do sẽ càng nhiều, đến một lúc nào đó cường độ huỳnh quang phát ra từ reporter đủ mạnh thì máy sẽ ghi nhận được tín hiệu huỳnh quang phát ra từ ống phản ứng khi nhận được nguồn sáng kích thích.
Reporter và quencher của Taqman probe
Như đã nói ở trên, reporter là một chất gắn vào đầu 5’ của Taqman probe, có khả năng phát được huỳnh quang khi nhận được nguồn sáng kích thích. Có nhiều loại hóa chất được sử dụng làm reporter. Trình bày trong bảng 6 là các hóa chất này với các chi tiết cần thiết như độ dài sóng của nguồn sáng kích thích, độ dài sóng của huỳnh quang phát ra, và các quencher tương ứng.
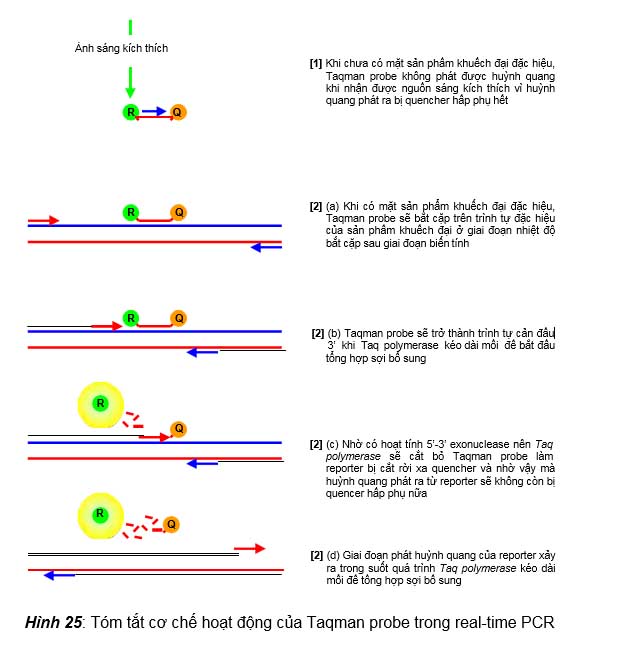
Quencher là chất có
khả năng hấp phụ được ánh sáng huỳnh quang phát ra từ reporter khi reporter nhận được ánh sáng kích thích. Có hai loại quencher, đó là quencher phát huỳnh quang và quencher phát nhiệt. Quencher phát huỳnh quang là quencher khi hấp phụ ánh sáng huỳnh quang từ reporter sẽ
- Khi chưa có mặt sản phẩm khuếch đại đặc hiệu, Taqman probe không phát được huỳnh quang khi nhận được nguồn sáng kích thích vì huỳnh quang phát ra bị quencher hấp phụ hết
- (a) Khi có mặt sản phẩm khuếch đại đặc hiệu, Taqman probe sẽ bắt cặp trên trình tự đặc hiệu của sản phẩm khuếch đại ở giai đoạn nhiệt độ bắt cặp sau giai đoạn biến tính
[2] (b) Taqman probe sẽ trở thành trình tự cản đầu
3’ khi Taq polymerase kéo dài mồi để bắt đầu
tổng hợp sợi bổ sung
[2] (c) Nhờ có hoạt tính 5’-3’ exonuclease nên Taq
polymerase sẽ cắt bỏ Taqman probe làm
reporter bị cắt rời xa quencher và nhờ vậy mà huỳnh quang phát ra từ reporter sẽ không còn bị quencer hấp phụ nữa
R
Q
chuyển năng lượng hấp
phụ được thành ánh sáng,
[2] (d) Giai đoạn phát huỳnh quang của reporter xảy ra trong suốt quá trình Taq polymerase kéo dài mồi để tổng hợp sợi bổ sung
do vậy quencher sẽ phát ra
Hình 25: Tóm tắt cơ chế hoạt động của Taqman probe trong real-time PCR
huỳnh quang nhưng không đến được CCD hay cảm biến quang vì bị cản lại bởi kính lọc chỉ dành cho ánh sáng huỳnh quang phát ra từ reporter. TAMRA là một ví dụ minh họa cho quencher phát huỳnh quang, thường được dùng cho taqman probe có reporter là FAM (bảng 6). Dùng quencher phát huỳnh quang sẽ có một trở ngại là huỳnh quang phát ra từ quencher có thể trùng với phổ huỳnh quang của reporter của một Taqman probe khác, nếu real-time PCR được thiết kế multiplex, do vậy mà khi chọn lọc reporter cho probe thứ 2 hay thứ 3 thì nhà nghiên cứu phải để ý để tránh trở ngại này. Quencher phát nhiệt là quencher sẽ chuyển năng lượng hấp phụ được thành nhiệt, ví dụ DABCYL hấp phụ huỳnh quang bước sóng 425, BHQ0 hấp thụ huỳnh quang bước sóng 430-520 (tối ưu 493), BHQ1 hấp thụ huỳnh quang 480-580 (tối ưu 534), BHQ2 hấp thụ huỳnh quang 560-670 (tối ưu 579), BHQ3 hấp thụ huỳnh quang 620-730 (tối ưu 672). Quencher phát nhiệt hiện nay được ưa chuộng hơn quencher phát huỳnh quang vì khả năng hấp phụ huỳnh quang rất mạnh, các BHQ được xem như các lỗ đen (black hole quencer, BHQ) hút lấy tất cả các huỳnh quang từ reporter không cho bất cứ huỳnh quang nào phát ra được từ reporter, đồng thời rất dễ dàng để thiết kế các taqman probe dùng cho multiplex PCR mà không lo đến phổ huỳnh quang của các reporter có bị trùng với phổ huỳnh quang của quencher vì quencher không hề phát huỳnh quang.
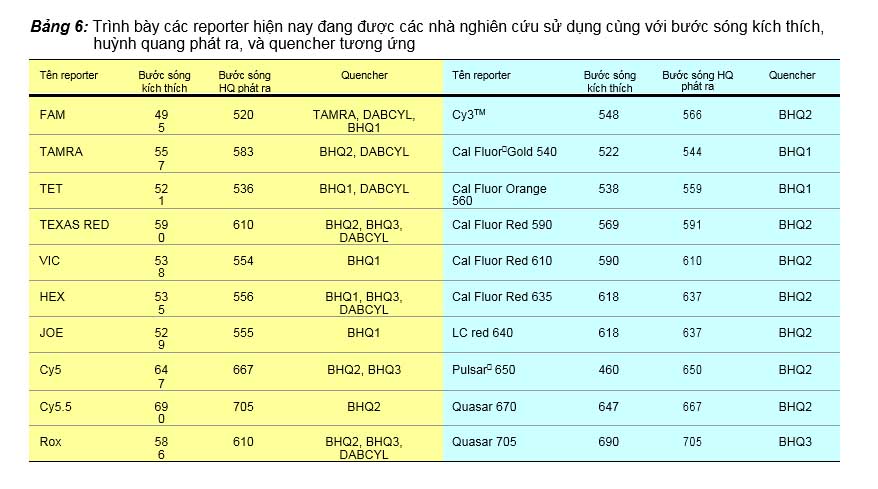
Bảng 6: Trình bày các reporter hiện nay đang được các nhà nghiên cứu sử dụng cùng với bước sóng kích thích, huỳnh quang phát ra, và quencher tương ứng
Tên reporter | Bước sóng kích thích | Bước sóng HQ phát ra | Quencher | Tên reporter | Bước sóng kích thích | Bước sóng phát ra | HQ | Quencher |
FAM | 495 | 520 | TAMRA, DABCYL, BHQ1 | Cy3TM | 548 | 566 | BHQ2 | |
TAMRA | 557 | 583 | BHQ2, DABCYL | Cal FluorGold 540 | 522 | 544 | BHQ1 | |
TET | 521 | 536 | BHQ1, DABCYL | Cal Fluor Orange 560 | 538 | 559 | BHQ1 | |
TEXAS RED | 590 | 610 | BHQ2, BHQ3, DABCYL | Cal Fluor Red 590 | 569 | 591 | BHQ2 | |
VIC | 538 | 554 | BHQ1 | Cal Fluor Red 610 | 590 | 610 | BHQ2 | |
HEX | 535 | 556 | BHQ1, BHQ3, DABCYL | Cal Fluor Red 635 | 618 | 637 | BHQ2 | |
JOE | 529 | 555 | BHQ1 | LC red 640 | 618 | 637 | BHQ2 | |
Cy5 | 647 | 667 | BHQ2, BHQ3 | Pulsar 650 | 460 | 650 | BHQ2 | |
Cy5.5 | 690 | 705 | BHQ2 | Quasar 670 | 647 | 667 | BHQ2 | |
Rox | 586 | 610 | BHQ2, BHQ3, DABCYL | Quasar 705 | 690 | 705 | BHQ3 | |
Thiết kế mồi và Taqman probe
Thiết kế mồi cho real-time PCR dùng Taqman probe cũng theo các qui luật thiết kế mồi chung cho PCR (trình bày trong phần thiết kế mồi dành real-time PCR dùng màu huỳnh quang chèn). Trong real-time PCR dùng Taqman probe thì nên thiết kế mồi có nhiệt độ chảy khoảng 55oC – 60oC và thấp hơn nhiệt độ chảy của Taqman probe
khoảng 5oC – 10oC. Để thiết kế Taqman probe, nên thiết kế để không dài quá 30 bases, tỷ lệ GC trong probe khoảng 30-80 % với C nhiều hơn G, vị trí bắt cặp của đầu 5’ của Taqman probe chỉ 2-5 bases cách vị trí 3’ của mồi bắt cặp trên cùng sợi đích, và rất quan trọng là đầu 5’ gắn reporter không phải là G vì G có thể là quencher cho rất nhiều reporter. Có thể sử dụng phần mềm Beacon Designer để thiết kế mồi và Taqman probe. Phần mềm này được cấp miễn phí cho các phòng thí nghiệm mua máy PCR iCycler cua Biorad. Ngoài ra, việc chọn reporter và quencher cũng là một vấn đề hết sức quan trọng. Xin tham khảo bảng 6 để có nhiều khả năng lựa chọn cho phù hợp.
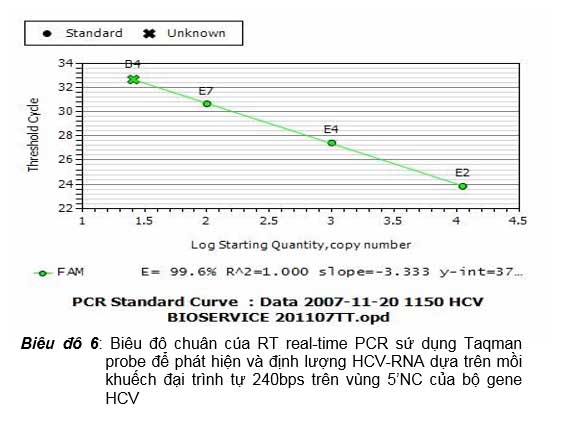
Khi thiết kế mồi cho real-time PCR dùng Taqman probe thì cũng nên lưu ý để chiều dài sản phẩm khếch đại trong khoảng 75-150 bps, không nên quá 150 bps để hiệu quả PCR có thể đạt được tối hảo. Tuy nhiên trên thực tế cũng còn tùy thuộc vào DNA đích có cho phép chúng ta chọn được mồi đạt được tối hảo để có sản phẩm khuếch đại đạt như vậy hay không. Công ty Nam Khoa đã có bộ thử nghiệm RT real-time PCR sử dụng Taqman probe để phát hiện và định lượng HCV-RNA dựa trên sự khuếch đại sợi cDNA đích dài 240 bps phiên mã ngược từ vùng 5’NC của bộ gene của virus mà vẫn đạt được hiệu quả PCR tối hảo thể hiện qua E% luôn đạt 90-105 % (biểu đồ 6); cũng chính nhờ vậy mà công ty triển khai được thử nghiệm giải trình tự sản phẩm real-time PCR này để xác định được genotype của HCV nhiễm trên bệnh nhân.
Chương trình luân nhiệt real-time PCR dùng taqman probe thường chỉ có hai giai đoạn nhiệt độ là: biến tính 94 – 95oC trong 15 – 30 giây, kế đó là giai đoạn vừa bắt cặp vừa kéo dài ở 60oC trong 30-60 giây và thiết bị
real-time sẽ hoạt động ở giai đoạn này. Ở giai đoạn nhiệt độ 60oC thì Taqman probe
Biểu đồ 6: Biểu đồ chuẩn của RT real-time PCR sử dụng Taqman probe để phát hiện và định lượng HCV-RNA dựa trên mồi khuếch đại trình tự 240bps trên vùng 5’NC của bộ gene HCV
sẽ bắt cặp vào sợi đích trước vì đây là nhiệt độ bắt cặp tối hảo của Taqman probe (thấp hơn Tm của Taqman probe là 65 – 70oC, nhưng bằng hay cao hơn Tm của mồi là 55oC
– 60oC) rồi sau đó mồi mới bắt cặp vào. Chính nhờ vậy mà khi Taq polymerase tổng hợp sợi bổ sung, enzyme này mới có cơ hội thủy giải được Taqman probe cản đầu nó. Nếu mồi bắt cặp trước vào sợi đích thì sẽ không có cơ hội để Taqman probe bắt cặp vào sợi đích và như vậy thì Taqman probe sẽ không thể bị thủy giải khi enzyme Taq polymerase tổng hợp sợi bổ sung. Đây chính là lý do giải thích tại sao phải thiết kế mồi có Tm khoảng 55-60oC và thấp hơn Tm của Taqman probe 5oC – 10oC.
Pha PCR mix cho real-time PCR dùng Taqman probe
Pha PCR mix cho real-time PCR dùng Taqman probe bao gồm các thành phần với các nồng độ hay hàm lượng như sau: (1) PCR buffer 1X, (2) Taq polymerase có hoạt tính 5’-3’ exonuclease 1.25 hàm lượng 2 unit cho một thể tích phản ứng, (3) dNTP 200
mM/each, (4) mồi xuôi và mồi ngược hàm lượng 10-25 pm cho mỗi thể tích phản ứng,
(5) MgCl2 5 mM, (6) Taqman probe 2-5 pm cho mỗi thể tích phản ứng; (7) Ngoài ra có thể thêm dUTP, UNG với nồng độ hay hàm lượng như các pha các PCR mix real-time hay PCR mix không real-time khác, và (8) màu huỳnh quang nền nếu thiết bị real-time yêu cầu. Nên thăm dò hàm lượng của Taqman probe để có cường độ huỳnh quang được ghi nhận là cao nhất, không phải cho hàm lượng của Taqman probe cao thì sẽ làm cường độ huỳnh quang phát ra sẽ cao mà có khi có tác động ngược lại vì sẽ làm các phân tử Taqman probe quá gần nhau nên huỳnh quang phát ra từ reporter của probe bị thủy giải vẫn bị các quencher của các Taqman probe khác hấp phụ.
Chọn reporter của Taqman probe cho multiplex PCR
Để thiết kế được các Taqman probe dùng cho multiplex PCR với các quencher là BHQ thì yếu tố quan trọng nhất là phải lựa chọn như thế nào để bước sóng kích thích các reporter, và nhất là bước sóng huỳnh quang phát ra từ các reporter phải cách xa nhau để tín hiệu huỳnh quang được thiết bị ghi nhận cho từng reporter sẽ không bị ảnh hưởng lên nhau. Ngoài ra trên thực tế sự lựa chọn reporter là còn tùy thuộc vào các kênh lọc màu có trên thiết bị real-time nữa. Biểu đồ 7 minh họa một lựa chọn được xem là tối ưu cho multiplex 5 màu thực hiện với real-time PCR trên iQ5 của Biorad. Trong sự lựa chọn này, chúng ta thấy bước sóng huỳnh quang tối đa của tất cả 5 reporter là cách xa nhau nhất và tất cả đều phù hợp với 5 kênh kính lọc nguồn sáng kích thích đến và huỳnh quang phát ra từ reporter (trình bày trong bảng 7).
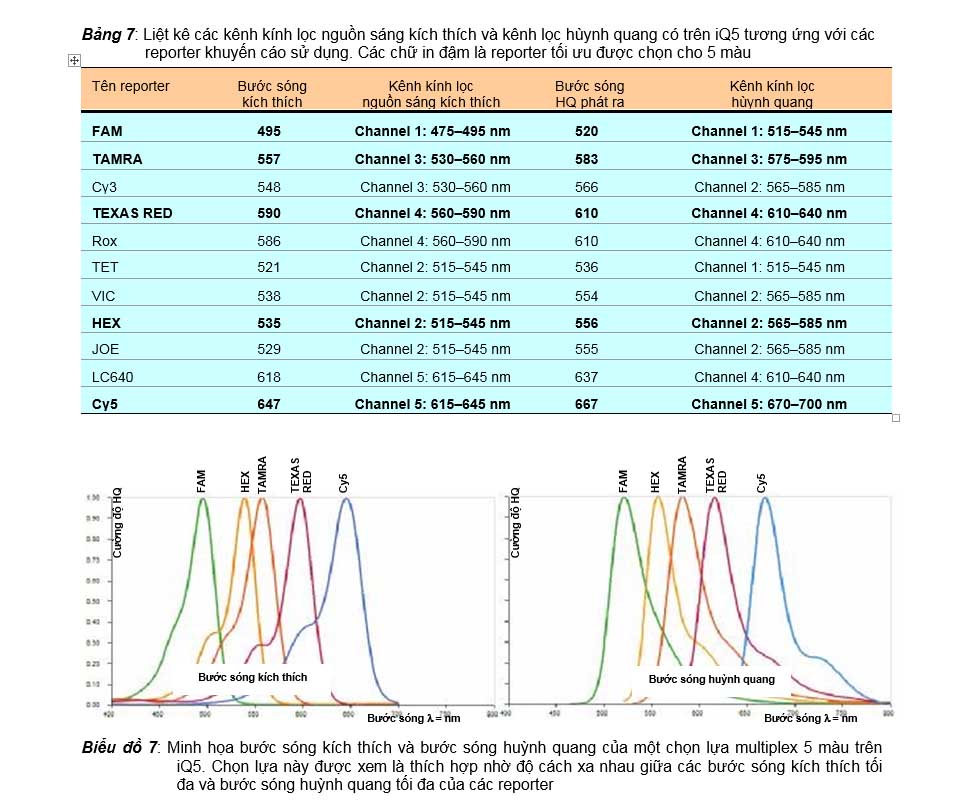
Bảng 7: Liệt kê các kênh kính lọc nguồn sáng kích thích và kênh lọc hùynh quang có trên iQ5 tương ứng với các reporter khuyến cáo sử dụng. Các chữ in đậm là reporter tối ưu được chọn cho 5 màu
Tên reporter | Bước sóng kích thích | Kênh kính lọc nguồn sáng kích thích | Bước sóng HQ phát ra | Kênh kính lọc hùynh quang |
FAM | 495 | Channel 1: 475–495 nm | 520 | Channel 1: 515–545 nm |
TAMRA | 557 | Channel 3: 530–560 nm | 583 | Channel 3: 575–595 nm |
Cy3 | 548 | Channel 3: 530–560 nm | 566 | Channel 2: 565–585 nm |
TEXAS RED | 590 | Channel 4: 560–590 nm | 610 | Channel 4: 610–640 nm |
Rox | 586 | Channel 4: 560–590 nm | 610 | Channel 4: 610–640 nm |
TET | 521 | Channel 2: 515–545 nm | 536 | Channel 1: 515–545 nm |
VIC | 538 | Channel 2: 515–545 nm | 554 | Channel 2: 565–585 nm |
HEX | 535 | Channel 2: 515–545 nm | 556 | Channel 2: 565–585 nm |
JOE | 529 | Channel 2: 515–545 nm | 555 | Channel 2: 565–585 nm |
LC640 | 618 | Channel 5: 615–645 nm | 637 | Channel 4: 610–640 nm |
Cy5 | 647 | Channel 5: 615–645 nm | 667 | Channel 5: 670–700 nm |
Biểu đồ 7: Minh họa bước sóng kích thích và bước sóng huỳnh quang của một chọn lựa multiplex 5 màu trên iQ5. Chọn lựa này được xem là thích hợp nhờ độ cách xa nhau giữa các bước sóng kích thích tối đa và bước sóng huỳnh quang tối đa của các reporter
- Real-time PCR sử dụng Beacon probe
Trong kỹ thuật này, probe được sử dụng có tên là Beacon molecular probe. Đây là probe có trình tự dài khoảng 25-40 bases, có đầu 5’ gắn với reporter và đầu 3’ gắn với quencher. Probe có trình tự 15-39 bases ở giữa là bổ sung với một trình tự đặc hiệu trên một sợi của DNA đích, còn hai đầu của probe thì mỗi đầu có một trình tự 5-6 bases bổ sung với nhau làm cho probe tạo thành cấu trúc hình kẹp tóc do trình tự của hai đầu bắt cặp nhau. Cơ chế hoạt động của Beacon probe được minh họa trong hình 26, tóm tắt như sau: (1) Ở giai đoạn nhiệt độ biến tính, cấu trúc kẹp tóc của Beacon probe không còn nên nếu ở giai đoạn này reporter nhận nguồn sáng kích thích thì nó sẽ phát huỳnh quang. (2) Ở giai đoạn nhiệt độ bắt cặp, nếu chưa có mặt sản phẩm khuếch đại đặc hiệu, Beacon probe sẽ không phát được huỳnh quang khi nhận được nguồn sáng kích
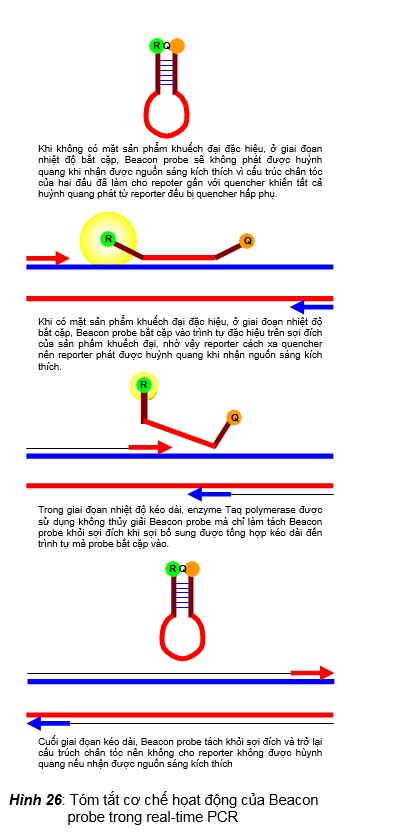
thích vì cấu trúc kẹp tóc của hai đầu đã làm cho reporter gần với quencher khiến tất cả huỳnh quang phát từ reporter đều bị quencher hấp phụ. Nhưng nếu có mặt sản phẩm khuếch đại đặc hiệu, Beacon probe sẽ bắt cặp vào trình tự đặc hiệu trên sợi đích của sản phẩm khuếch đại, nhờ vậy reporter cách xa quencher nên reporter phát được huỳnh quang khi nhận nguồn sáng kích thích. (3) Trong giai đoạn nhiệt độ kéo dài, enzyme Taq polymerase không thủy giải Beacon probe mà chỉ làm tách Beacon probe khỏi sợi đích khi sợi bổ sung được tổng hợp kéo dài đến trình tự mà probe bắt cặp vào. Cuối giai đoạn kéo dài, Beacon probe tách khỏi sợi đích và trở lại cấu trúc kẹp tóc nên không cho reporter phát được huỳnh quang nếu nhận được nguồn sáng kích thích.
Beacon probe có một số ưu điểm, đó là: (1) rất đặc hiệu vì chỉ cần trình tự sợi khuôn khác biệt là sẽ không có bắt cặp và như vậy là sẽ không có huỳnh quang –
Khi không có mặt sản phẩm khuếch đại đặc hiệu, ở giai đọan nhiệt độ bắt cặp, Beacon probe sẽ không phát được huỳnh quang khi nhận được nguồn sáng kích thích vì cấu trúc chân tóc của hai đầu đã làm cho repoter gần với quencher khiến tất cả huỳnh quang phát từ reporter đều bị quencher hấp phụ.
Khi có mặt sản phẩm khuếch đại đặc hiệu, ở giai đoạn nhiệt độ bắt cặp, Beacon probe bắt cặp vào trình tự đặc hiệu trên sợi đích của sản phẩm khuếch đại, nhờ vậy reporter cách xa quencher nên reporter phát được huỳnh quang khi nhận nguồn sáng kích thích.
Trong giai đọan nhiệt độ kéo dài, enzyme Taq polymerase được sử dụng không thủy giải Beacon probe mà chỉ làm tách Beacon probe khỏi sợi đích khi sợi bổ sung được tổng hợp kéo dài đến trình tự mà probe bắt cặp vào.
Cuối giai đọan kéo dài, Beacon probe tách khỏi sợi đích và trở lại cấu trúch chân tóc nên không cho reporter không được hùynh quang nếu nhận được nguồn sáng kích thích
Hình 26: Tóm tắt cơ chế họat động của Beacon probe trong real-time PCR
chính nhờ vậy Beacon rất ưu việt để phát hiện SNP; (2) rất tốt để thực hiện multiplex phát hiện cùng lúc nhiều tác nhân đích. Tuy nhiên, Beacon tương đối khó thiết kế, nhất là khi thiết kế trình tự ở hai đầu 5’ và 3’ phải chú ý để có sự bắt cặp đủ mạnh làm cho probe có cấu trúc kẹp tóc mà không làm cho cấu trúc của probe tự gấp lại thành cấu hình không kẹp tóc, vì như vậy sẽ làm cho huỳnh quang của reporter phát ra tự do không mong muốn. Ngoài ra sự bắt cặp của trình tự ở hai đầu cũng không được quá mạnh để không ngăn cản probe bắt cặp vào sợi đích khi có mặt của sợi đích. Chương trình Beacon designer được cung cấp miễn phí khi mua iCycler của Biorad là một
trong các chương trình rất tốt, giúp người sử dụng dễ dàng thiết kế Beacon probe trên trình tự sợi khuôn đã nhập vào.
- Real-time PCR sử dụng probe lai (hybridization probes)
Trong kỹ thuật này, người ta dùng 2 probe. Probe
lai 1 có đầu 3’ gắn một chất phát huỳnh quang D (donnor), và probe lai 2 có đầu 5’ gắn một chất phát huỳnh quang A (acceptor). Để tránh không cho probe lai 2 này có thể kéo dài khi nó bắt cặp lên sợi khuôn, người ta thường gắn thêm một gốc phosphate ở đầu 3’. Hai probe lai này được thiết kế sao cho khi bắt cặp trên sợi khuôn thì đầu 3’ của probe lai 1 chỉ cách đầu 5’ của probe lai 2 khoảng từ 3-5 bases, ngoài ra phải chọn D và A sao cho huỳnh quang từ D phát ra có độ dài sóng trùng với nguồn sáng kích thích của A để làm cho A phát ra được huỳnh quang có độ dài sóng khác biệt với huỳnh quang phát ra từ D. Để hệ thống hoạt động được thì thiết bị realtime phải phát được nguồn sáng kích thích D, nhưng CCD hay cảm quang phải có kính lọc để chỉ ghi nhận huỳnh quang phát ra từ A chứ không phải từ D. Cơ chế hoạt động của hybridization probes được tóm tắt như sau (hình 27): Ở giai đoạn nhiệt độ bắt cặp, nếu không có sản phẩm khuếch đại đặc hiệu thì
Khi chưa có mặt sản phẩm khuếch đại đặc hiệu, huỳnh quang phát ra từ D của probe lai 1 không kích thích được A của probe lai 2 vì chúng ở cách xa nhau
Khi có mặt sản phẩm khuếch đại đặc hiệu, Probe lai 1 và probe lai 2 bắt cặp lên sợi khuôn làm cho D rất gần A nên huỳnh quang phát ra từ D (có bước sóng trùng với bước sóng nguồn sáng kích thích của A) sẽ được A hấp phụ rồi chuyển sang phát huỳnh quang với bước sóng khác biệt với bước sóng phát huỳnh quang của D
Trong giai đoạn kéo dài, Taq polymerase vì không có hoạt tính 5’-3’ exonuclease nên sẽ tách các probe lai 1 rồi probe lai 2 khỏi sợi khuôn, do vậy là A tách rời D nên D không còn nhận được buồn sáng kích thích từ D nữa và A sẽ không còn phát huỳnh quang được.
Hình 27:
Tóm tắt cơ chế hoạt động của probe lai trong real-time PCR
D của probe lai 1 phát huỳnh quang do nhận được nguồn sáng kích thích, nhưng thiết bị real-time không ghi nhận được huỳnh quang này vì bước sóng phát ra từ D không đi qua được kính lọc để đến CCD camera hay cảm quang. Nếu có sản phẩm khuếch đại hiện diện thì probe lai 1 và probe lai 2 bắt cặp được vào sợi khuôn, do vậy mà huỳnh quang phát ra từ D sẽ là nguồn sáng kích thích A, làm cho A phát huỳnh quang và thiết bị real-time sẽ ghi nhận được huỳnh quang này nếu lượng sản phẩm khuếch đại đạt đủ ngưỡng để huỳnh quang phát ra từ A qua được kính lọc để đến được CCD camera hay cảm quang của thiết bị real-time. Cơ chế hoạt động của probe lai như vậy nên kỹ thuật này còn gọi là kỹ thuật FRET real-time (Fluorescence Resonance Energy Transfer).
Cũng như tất cả các cơ chế phát huỳnh quang khác của real-time PCR, kỹ thuật real-time PCR sử dụng các probes lai được ứng dụng để định lượng tác nhân đích có trong mẫu. Ngoài áp dụng này, real-time PCR sử dụng các probe lai còn có một ưu điểm, đó là ứng dụng để định genotype của tác nhân đích hay là định được sự khác biệt chỉ một nucleotide (SNP) của các tác nhân đích. Nguyên tắc của ứng dụng này là nhiệt độ chảy của hai probe lai trên các sơi khuôn khác biệt về trình tự dù chỉ một nucleotide cũng sẽ khác nhau, nên làm cho đường biểu diễn đỉnh chảy của chúng cũng khác nhau. Hình 28 và biểu đồ 8 là một minh họa để hiểu rõ hơn tại sao real-time PCR với probes lai có thể giúp phân biệt được các khác biệt của tác nhân đích về genotype hay thậm chí chỉ một SNP. Để có thể thực hiện được ứng dụng này thì người làm thí nghiệm sẽ chạy thêm một chương trình phân tích melt-curve sau khi máy đã hoàn tất chương trình luân nhiệt real-time.
80oC ·
81oC ·
Sản phẩm khuếch đại B Sản phẩm khuếch đại A
Hình 28:
Minh họa cho thấy sản phẩm khuếch đại A chỉ khác B môt nucleotide nhưng nhiệt độ chảy của probe lai trên sợi khuôn đã khác biệt
78oC 79oC 80oC 81oC 82oC 83oC
Biểu đồ 8:
Minh họa cho thấy đỉnh nhiệt độ chảy của A phân biệt rõ với đỉnh nhiệt độ chảy của B. Nhờ vậy có thể thấy được A và B khác biệt nhau
- Các kỹ thuật real-time PCR sử dụng probe khác
Phần trên là giới thiệu tương đối chi tiết về 3 phương pháp real-time PCR sử dụng probes làm chất phát huỳnh quang. Đây là những phương pháp thường được sử dụng trong nhiều nghiên cứu hay là trong chẩn đoán lâm sàng. Ngoài 3 phương pháp dùng probe làm chất phát huỳnh quang này, còn có nhiều phương pháp dùng probes hay cả primer nữa để làm chất phát huỳnh quang, như Eclipse probe, Amplifluor primers, Scorpion primers, Lux primers, BD-QZyme primers…Xin tham khảo tài liệu Real-time
PCR Applications Guide (http://www.gene-quantification.de/real-time-pcr-guide-bio– rad.pdf) để rõ hơn.
Các ứng dụng của real-time PCR
1. Định lượng tác nhân đích có trong mẫu thử
Đặc trưng của real-time PCR là phát hiện sản phẩm khuếch đại trong quá trình chạy PCR khi sản phẩm khuếch đại từ DNA đích được nhân bản đạt đủ số lượng để làm cho ống phản ứng phát được huỳnh quang khi nhận được nguồn sáng kích thích. Chính nhờ đặc trưng này mà người làm thí nghiệm có thể biết được số lượng bản DNA đích ban đầu có trong ống phản ứng dựa vào sự xuất hiện huỳnh quang của ống phản ứng sớm hay muộn, tức là chu kỳ ngưỡng (Ct) của ống phản ứng nhỏ hay lớn. Có hai cách định lượng tác nhân đích dựa vào mục đích định lượng mà người làm thử nghiệm quan tâm:
- Định lượng tuyệt đối
Người làm thử nghiệm sẽ sử dụng định lượng tuyệt đối nếu mục đích của xét nghiệm là định lượng để biết rõ số copies của tác nhân đích có trong mẫu thử hay trong bệnh phẩm. Ví dụ xét nghiệm định lượng virus viêm gan B, hay viêm gan C, hay HIV trong mẫu máu của bệnh nhân để theo dõi hiệu quả điều trị đặc hiệu. Phương pháp định lượng này đòi hỏi phải biết rõ lượng (thể tích, hay có thể là trong lượng) của mẫu thử. Để có thể thực hiện phương pháp định lượng tuyệt đối, nhà nghiên cứu hay người làm thí nghiệm phải thực hiện xét nghiệm real-time PCR của mẫu thử cùng lúc với các mẫu chuẩn đã biết trước số lượng rồi tính ra số copies DNA của tác nhân đích có trong ống
Biểu đồ 9: Biểu đồ chuẩn là một yếu tố quan trọng để làm được định lượng tuyệt đối. Nhờ biểu đồ chuẩn mà chương trình của máy có thể tính được hàm lượng DNA đích ban đầu có trong ống phản ứng.
phản ứng dựa vào đường biển diễn chuẩn (minh họa ở biểu đồ 9) xác định mối quan hệ giữa chu kỳ ngưỡng (Ct) với số lượng copies DNA đích ban đầu có trong ống phản ứng. Cuối cùng xác định số copies tác nhân đích có trong mẫu thử hay bệnh phẩm dựa vào hệ số pha loãng mẫu và hệ số tách chiết DNA hay RNA của phương pháp chuẩn bị mẫu trước khi thực hiện PCR.
Cụ thể phương pháp tính toán số copies của DNA đích ban đầu có trong ống phản
ứng dựa vào đường biểu diễn chuẩn như sau:
- Đường biểu diễn chuẩn cho được một hàm số biểu thị mối tương quan giữa chu kỳ ngưỡng (Y = Ct) với log10 của số lượng bản DNA đích ban đầu có trong ống phản ứng (X = log10 Sq). Hàm số đó là: Y = [slope (X)] + intercept, và các thông số slope và intercept đều hiển thị trên biểu đồ chuẩn.
- Từ hàm số này, người làm thí nghiệm sẽ hiểu được tại sao máy tính hiển thị được Sq, đó là nhờ tính toán từ Sq = 10[(Ct – intercept)/slope].
- Định lượng tương đối
Nếu muốn định lượng tác nhân đích mà người làm thí nghiệm không thể cân đo đong đếm được mẫu thử để có được số lượng chính xác thì nên làm phương pháp định lượng tương đối. Ví dụ xác định tôm sú bị nhiễm một loại virus gây bệnh nào đó nhiều hay ít; trong trường hợp này người làm thí nghiệm đâu có thể cân chính xác số lượng tôm post được nghiền để thử nghiệm do vậy mà nếu dùng phương pháp định lượng tuyệt đối sẽ gặp trở ngại vì nếu lượng tôm thử nghiệm nhiều quá thì lượng nhiễm virus dù ít vẫn có thể cho con số định lượng tuyệt đối cao và ngược lại nếu lượng tôm thử nghiệm quá ít thì con số định lượng tuyệt đối có thể thấp dù tôm nhiễm virus cao. Định lượng tương đối cũng được sử dụng trong các nghiên cứu về ung thư để định lượng một biển hiện gene nào đó, hay cũng được sử dụng trong định lượng sản phẩm biến đổi gene. Các ví dụ minh họa trong các phương pháp định lượng tương đối sau đây sẽ làm rõ hơn về định lượng tương đối.
Định lượng tương đối dựa trên đơn vi khối lượng (unit mass)
Phương pháp định lượng này có thể áp dụng trong định lượng biểu hiện gene với lượng mẫu thử nghiệm phải được xác định chính xác (lượng tế bào, lượng nucleic acid tách chiết từ mẫu…). Lấy ví dụ định lượng biểu hiện gene P53 trên cấy tế bào có thử
chất nghiên cứu X gây ung thư so với cấy tế bào chứng không thử chất nghiên cứu X. Nhà nghiên cứu thực hiện hai lô cấy tế bào cùng ngày tuổi, sau đó một lô thử thêm chất X, một lô chứng không thử chất X. Sau đó lấy cùng một lượng tế bào của cả hai lô để tách chiết và định lượng RNA toàn bộ của cả hai lô. Lấy cùng một lượng RNA tách chiết được của cả hai lô (ví dụ 50 ng cho mỗi lô), thực hiện thử nghiệm RT real-time PCR với mồi đặc hiệu mRNA của P53. Xác định Ct của mRNA của P53 với giá trị Ct[T] là chu kỳ ngưỡng của khuếch đại mRNA của P53 trên 50 ng RNA tách chiết từ lô tế bào có thử chất X và Ct[C] là chu kỳ ngưỡng của khuếch đại mRNA của P53 trên 50 ng RNA tách chiết từ lô tế bào chứng không có thử chất X. Nếu hiệu quả PCR là đạt 100 %, nghĩa là cứ sau mỗi chu kỳ nhiệt lượng DNA luôn tăng gấp đôi (E = 2) thì tỷ lệ biểu hiện gene P53 của lô tế bào có thử chất X so với lô chứng sẽ là:
Tỷ lệ biểu hiện gene P53 [Thử/Chứng] = 2(Ct[C]-Ct[T])
Ví dụ, nếu Ct[T] là 12, Ct[C] là 15, thì tỷ lệ biểu hiện gene P53 của tế bào có thử chất X so với tế bào không thử chất X là 2(15-12) = 23 = 8. Có nghĩa là chất X đã làm cho biểu hiện P53 trên tế bào tăng gấp 8 lần so với chứng.
Định lượng tương đối dựa trên gene tham chiếu (reference gene)
Phương pháp định lượng này có thể áp dụng trong định lượng tác nhân gây bệnh trong các mẫu thử mà không thể xác định chính xác số lượng, hay là định lượng biểu hiện gene trong các mô ung thư. Có thể áp dụng một trong các phương pháp sau đây:
Phương pháp 2–DDCt của Livak
Phương pháp này có thể dùng để nghiên cứu biểu hiện một gene trong mẫu cấy tế bào ung thư, so với mẫu cấy tế bào bình thường; hay cũng có thể nghiên cứu biểu hiện một gene nào đó trên cấy tế bào có thử chất gây ung thư X so với cấy tế bào không thử X như ví dụ trên. Trong phương pháp này, người làm thí nghiệm sẽ định lượng không chỉ gene cần nghiên cứu (Target = Tg) mà cả một gene tham chiếu (reference = Ref) tức là gene mà các nghiên cứu trước đó đã xác định là có biểu hiện như nhau giữa hai mẫu tế bào (Thử: tế bào ung thư hay có thử chất X, gọi là mẫu T; và Chứng: tế bào không ung thư hay không thử chất X, gọi là mẫu C). Như vậy ứng với mẫu T và mẫu C sẽ có các kết quả định lượng cho cả gene Tg và gene Ref qua các thông số:
- Ct(T/Tg): là chu kỳ ngưỡng của gene đích (Tg) trong mẫu thử (T),
- Ct(T/Ref): là chu kỳ ngưỡng của gene tham chiếu (Ref) trong mẫu thử (T),
- Ct(C/Tg): là chu kỳ ngưỡng của gene đích (Tg) trong mẫu chứng (C), và
- Ct(C/Ref): là chu kỳ ngưỡng của gene tham chiếu (Ref) trong mẫu chứng (C).
Dựa trên các thông số này, chúng ta sẽ thường hóa (normalized) chu kỳ ngưỡng của gene đích trên mẫu thử (T) và mẫu chứng (C) bằng cách tính hiệu số chênh lệch Ct của gene đích với gene tham chiểu trên các mẫu:
Mẫu thử: | DCt(T) | = | Ct(T/Tg) – Ct(T/Ref) |
Mẫu chứng: | DCt(C) | = | Ct(C/Tg) – Ct(C/Ref) |
Sau đó sẽ thường hóa DCt mẫu thử bằng hiệu số chênh lệch DCt(T) với DCt(C)
DDCt = DCt(T) – DCt(C)
Từ đó tính ra tỷ lệ biểu hiện của gene đích trên mẫu thử so với mẫu chứng, nếu hiệu quả PCR của cả hai đều đạt 100%, là R = 2–DDCt
Ví dụ sau đây minh họa cho phương pháp 2DDCt của Livak áp dụng trong nghiên cứu biểu hiện gene P53 trên cấy tế bào buồng trứng ung thư (T) so với tế bào buồng trứng bình thường (C). Cả hai đều được tách chiết RNA toàn bộ rồi lấy 50 ng RNA được tách chiết để làm thử nghiệm RT real-time PCR với 2 Taqman probe và mồi đặc hiệu cho mRNA của P53 và của gene tham chiếu là GAPDH (các nghiên cứu trước đó chứng minh là GAPDH biểu hiện không khác biệt trên mô bình thường và mô ung thư). Kết quả trình bày trong bảng 8 dưới đây:
Bảng 8: Chu kỳ ngưỡng của gene P53 (Tg) và GADPH (Ref) của hai mẫu cấy tế bào buồng trứng ung thư (T) và bình thường (C).
Mẫu cấy tế bào | Ct P53 | (Tg) | Ct GAPDH | (Ref) |
Bình thường (C) | 15.0 | 16.5 | ||
Ung thư (T) | 12.0 | 15.9 | ||
Với các kết quả trình bày trong bảng 8 này, chúng ta sẽ tính được
DCt(T) = Ct(T/Tg) – Ct(T/Ref) = 12.0 – 15.9
= -3.9
DCt(C) = Ct(C/Tg) – Ct(C/Ref) = 15.0 – 16.5
= -1.5
DDCt = DCt(T) – DCt(C) = -3.9 – (-1.5) = -2.4
Từ đó tính ra tỷ lệ biểu hiện của P53 trên mẫu thử so với mẫu chứng, nếu hiệu quả PCR của cả hai đều đạt 100%, là R = 2-(-2.4) = 5.3
Có nghĩa là P53 ở tế bào ung thư biểu hiện gấp 5.3 lần ở tế bào bình thường.
Phương pháp Pfaffl
Phương pháp 2DDCt của Livak áp dụng trên real-time PCR mà hiệu quả PCR của cả hai gene đích và tham chiếu đều đạt 100% tức là E = 2. Nếu hiệu quả PCR của hai gene khác nhau, gene đích (Tg) là E(Tg), và gene tham chiếu (Ref) là E(Ref) thì chúng ta phải sử dụng một cách tính tổng quát hơn của phương pháp Pfaffl để tính tỷ lệ (R) biểu hiện của gene đích trên mẫu thử so với mẫu chứng:
Công thức này thật ra chỉ là dạng tổng quát của công thức Livak, vì nếu cả hai gene đều có hiệu quả PCR 100%, tức E = 2, thì công thức trên sẽ là:
R = 2DCt(C/Tg-T/Tg)/2DCt(C/Ref-T/Ref) = 2DCt(C/Tg-T/Tg) – DCt(C/Ref-T/Ref)
= 2-[DCt(T/Tg-C/Tg)] – [DCt(T/Ref-C/Ref)] = 2-DDCt
Minh họa bằng cách áp dụng vào ví dụ trên, nếu cả hai gene đều có hiệu quả PCR là 100% (E = 2) thì công thức trên sẽ được tính là:
R = 215-12/216.5-15.9
= 23/20.6 = 8/1.52 = 5.3
Định lượng tương đối dựa trên khối lượng vật chủ
Để có thể định lượng được tỷ lệ nhiễm tác nhân virus gây bệnh trên tôm nhiều hay ít thì phải xác định được lượng mẫu được thử nghiệm. Do vậy cần phải có hai hệ thống, một là hệ thống thử để định lượng được tác nhân virus gây bệnh có trong mẫu thử, và một là hệ thống tham chiếu định lượng được lượng mẫu đưa vào thử nghiệm. Với phương pháp real-time PCR thì thông số định lượng sẽ có được chính là chu kỳ ngưỡng (Ct), và tỷ lệ nhiễm tác nhân virus gây bệnh sẽ chính là tỷ lệ % Ct của đường biểu diễn khuếch đại của một đọan gene đặc hiệu tác nhân đích (gọi là Ct[T]) so với Ct của đường biểu diễn khuếch đại của một đọan gene giữ nhà hiện diện trong bộ gene vật chủ (gọi là Ct[R]). Chúng tôi đặt tên tỷ lệ này là IP (Infected pathogen).
Đối với hệ thống thử thì nguyên tắc của kỹ thuật real-time PCR là khuếch đại và
đồng thời phát hiện một đọan DNA đặc hiệu từ bộ gene của tác nhân virus gây bệnh
nhờ sử dụng một cặp mồi đặc hiệu và một Taqman probe đặc hiệu có gắn chất phát hùynh quang FAM ở đầu 5’ và chất hấp phụ hùynh quang ở đầu 3’. Trong khi chạy PCR, một khi có sản phẩm khuếch đại đặc hiệu xuất hiện trong ống PCR thì taqman probe đặc hiệu với trình tự đích sẽ bắt cặp vào sản phẩm khuếch đại và sẽ bị thủy giải bởi men taq polymerase (nhờ hoạt tính 5’- 3’ exonuclease) khi tổng hợp sợi bổ sung ở giai đọan kéo dài. Sự thủy giải taqman probe sẽ làm tách rời chất phát hùynh quang FAM ở đầu 5’ khỏi chất hấp phụ hùynh quang (quencher) ở đầu 3’ của probe, nhờ vậy ống phản ứng sẽ phát hùynh quang khi nhận được ánh sánh kích thích, và sự phát hùynh quang này sẽ được ghi nhận bởi đầu đọc real-time của máy. Mẫu thử chứa nhiều bản đích ban đầu sẽ có chu kỳ ngưỡng (Ct = chu kỳ mà đầu đọc realtime của máy bắt đầu ghi nhận được có tín hiệu hùynh quang trong ống phản ứng) phát hiện sớm hơn là mẫu thử có ít bản đích. Đối với hệ thống chỉ thị thì cũng tương tự như vậy nhưng mồi và Taqman probe thì sẽ được thiết kế để đặc hiệu cho một gene giữ nhà hiện diện trong bộ gene của vật chủ. Ở đây, để tránh sự cạnh tranh lẫn nhau giữa hai hệ thống thử và tham chiếu làm cho sự định lượng không chính xác, chúng tôi thiết kế hai hệ thống này trong hai ống phản ứng khác nhau, do vậy mà Taqman probe của hệ thống tham chiếu cũng sẽ sử dụng cùng màu phát hùynh quang FAM ở đầu 5’ giống như Taqman probe của hệ thống thử.
Ví dụ định lượng virus gây đỏ mang (GAV, Gill associated virus) nhiễm trong mẫu tôm bằng bộ thử nghiệm RT qPCR phát hiện và định lượng GAV do công ty Nam Khoa sản xuất. Nguyên tắc của bộ thử nghiệm này, là định lượng GAV đồng thời với định lượng gene tham chiếu là cytochrome b của tôm có trong mẫu để xác định được tỷ lệ tôm nhiễm bệnh. Phương pháp thực hiện là:
- Trước hết lấy một lượng tôm thử nghiệm không cần chính xác số lượng, thực hiện tách chiết RNA tòan phần rồi đưa vào tổng hợp cDNA với mồi ngẫu nhiên;
- Sau đó thực hiện real-time PCR sử dụng Taqman probe và mồi đặc hiệu tương ứng cho GAV và cho gene cytochrome b. Kết quả sẽ cho chúng ta Ct[GAV] của GAV và Ct[Cytob] của gene cytochrome b. Tỷ lệ Ct[GAV]/Ct[Cytob] cho biết tỷ lệ nhiễm GAV trên mẫu tôm.
2. Xác định tỷ lệ biến đổi gene (GMO, Gene Modified Organism) có trong mẫu
Lợi điểm chính yếu của sinh vật biến đổi gene chính là làm tăng giá trị dinh dưỡng và/hay kháng lại sâu bệnh hay thuốc diệt cỏ. Đậu, bắp, lúa mì, lúa và bông là các cây lương thực thường được biến đổi gene nhất. Tuy có lợi điểm nhưng mức độ an tòan vẫn chưa được biết rõ nên nhiều quốc gia đòi hỏi kiểm soát tỷ lệ biến đổi gene của sinh vật và sản phẩm của nó, và chính do vậy mà cần phải có các phương pháp chính xác và nhạy cảm để làm được việc này. Các phương pháp hiện nay đang được sử dụng là ELISA và PCR. ELISA tương đối rẽ và nhanh, nhưng phương pháp PCR có lợi điểm là nhạy cảm, và có thể định lượng được. Cơ sở của PCR là định lượng một promoter thường có trong vector để biểu hiện được gene đã chèn vào vector này sau khi chuyển gene vào sinh vật đích. Ví dụ đậu biến đổi gene để kháng được thuốc trừ cỏ là do nhận được gene giúp phục hồi một đường biến dưỡng acid amin thiết yếu vốn dĩ bị glyphosate phá hủy. Đây chính là gene enolpyruvylshikimate-3-phosphate synthase (epsps) được cô lập từ vi khuẩn có ở đất, đó là Agrobacterium sp. dòng cp4. Gene này được chèn vào vector là virus khảm súp-lơ (cauliflower mosaic virus, CaMV) có mang promoter 35S rồi chuyển vào cây đậu. Vector có mang promoter 35S này rất thường được dùng để chuyển gene trong thực vật là vì phổ chuyển gene và biểu hiện được gene rất rộng cho nhiều lọai cây. Ngoài ra promoter 35S có trình tự rất ổn định và được biết rõ. Chính vì vậy nên CaMV 35S promoter là đối tượng chính yếu dùng để định tính và định lượng tỷ lệ biến đổi gene trong các sinh vật hay sản phẩm biến đổi gene. Sau đây là một ví dụ định lượng tỷ lệ biến đổi gene trong đậu nành để minh họa.
Để thực hiện được thử nghiệm, trước hết phải có các mẫu chuẩn tức là mẫu đậu nành đã có trộn thêm đậu nành biến đổi gene theo tỷ lệ biết trước từ 0% đến 5% (mua từ Sigma). Gene đích để định lượng là promoter 35S, gene tham chiếu là gene lectin của đậu nành. Các mẫu chuẩn và mẫu thử đểu được tách chiết DNA (dùng bộ tách chiết DNA cho thực vật, như bộ DNeasy plant mini kit của Qiagen có thể tách chiết được 1-2mg từ 55-100mg sản phẩm). Thực hiện real-time PCR (sử dụng SYBR hay Taqman probe) định lượng hai gene trên một lượng nhất định DNA tách chiết được từ các mẫu thử và mẫu chuẩn. Kết quả Ct được ghi nhận và trình bày trong bảng 9. Biểu đồ 10 trình bày mối quan hệ tuyến tính giữa tỷ lệ GMO% và DCt.
Bảng 9: Chu kỳ ngưỡng của S35 và lectin của các mẫu chuẩn và của các mẫu thử
Mẫu và chuẩn | Ct(S35) | Ct(lectin) | DCt | |
Chuẩn GMO | 0.0% | ND | 24.40 | ND |
Chuẩn GMO 0.1% 35.96 24.30 11.66
Chuẩn GMO 0.5% 33.19 23.60 09.59 | ||||
Chuẩn GMO | 1.0% | 32.12 | 23.60 | 08.52 |
Chuẩn GMO | 2.0% | 31.34 | 24.00 | 07.34 |
Chuẩn GMO 5.0% 29.76 23.80 05.96
Mẫu 1 27.22 21.85 05.37 | ||||
Mẫu 2 | ND | 20.73 | ND | |
log(GMO%)
Biểu đồ 10: Biểu đồ chuẩn mối quan hệ giữa DCt và tỷ lệ GMO
Từ hàm số tương quan y = -0.295x + 1.592 với y là log(GMO%) và x là DCt, chúng ta sẽ tính được mẫu 1 với DCt là 05.37 thì tỷ lệ GMO% là 7.9%, còn của mẫu 2 là 0-0.1%.
3. Phát hiện khác biệt SNP và xác định kiểu di truyền
Sản phẩm khuếch đại của các trình tự đích có thể khác biệt chỉ một nucleotide, gọi là SNP, hay là có thể khác biệt một số nucleotide của một trình tự. Phát hiện các khác biệt này nhiều lúc rất có ý nghĩa, chẳn hạn phát hiện đột biến kháng thuốc là phát hiện các SNP, hay phân biệt các kiểu di truyền của các tác nhân đích có trong mẫu thử là các bệnh phẩm lấy từ bệnh nhân là phát hiện các trình tự khác biệt. Real-time PCR hoàn toàn có thể ứng dụng để phát hiện khác biệt SNP và xác định kiểu di truyền.
Với real-time PCR dùng màu huỳnh quang chèn làm chất phát huỳnh quang thì phải sử dụng màu HRM và phải thực hiện chương trình phân tích HRM sau khi hoàn tất chương trình luân nhiệt thì mới có thể phát hiện được khác biệt SNP và xác định được kiểu di truyền (xem phần kỹ thuật phân tích phân giải cao nhiệt độ chảy).
Với real-time PCR dùng probe làm chất phát huỳnh quang thì tùy sự tiện lợi hay quen thuộc với kỹ thuật nào mà nhà nghiên cứu hay người làm thí nghiệm có thể chọn phương pháp thích hợp với mình. Ví dụ với Taqman probe, để phát hiện sự khác biệt SNP và định genotype, nhà nghiên cứu phải thiết kế các Taqman probe (mang các chất phát huỳnh quang R khác nhau) bắt cặp trên cùng vị trí của sản phẩm khuếch đại nhưng các probe này có sự khác biệt về trình tự tương ứng với sự khác biệt SNP hay khác biệt trình tự giúp phân biệt genotype. Trong giai đoạn bắt cặp thì probe có trình tự
tương thích nhất với trình tự đích trên sản phẩm khuếch đại sẽ bắt cặp lên sợi đích và sẽ bị Taq polymerase thủy giải, do vậy đường biểu diễn khuếch đại tương ứng với màu huỳnh quang của probe sẽ ngóc lên và đến cực đại trong khi tín hiệu của các màu huỳnh quang khác sẽ vẫn nằm dưới huỳnh quang nền vì các probe tương ứng vẫn còn nguyên vẹn và không bị thủy giải. Hình 29 dưới đây minh họa cơ chế hoạt động của Taqman probe trong phát hiện SNP hay genotype.
Hình 29: Minh họa cơ chế họat động của Taqman probe trong phát hiện khác biệt SNP hay genotype với probe R1 cho wild type và probe R2 cho kiểu đột biến. Hình trên chỉ có wild type. Hình giữa chỉ có đột biến, và hình dưới là heterozygote vừa wild type, vừa đột biến
Phương pháp real-time PCR dùng probe lai (FRET) cũng được các nhà nghiên cứu sử dụng để phát hiện các khác biệt SNP và đặc biệt là khác biệt genotype. Phương pháp này tỏ ra ưu điểm hơn phương pháp sử dụng Taqman probe trong phát hiện các khác biệt genotype, nhất là khi các genotype của tác nhân đích ngoài khác biệt nhau về trình tự đặc trưng còn chứa các khác biệt trình tự do sự khác biệt dòng hay khác biệt vì đột biến trong quá trình phát triển (ví dụ HIV, HCV, HBV…). Lý do là rất khó thiết kế được Taqman probe đặc hiệu cho một genotype vì ngoài trình tự đặc hiệu cho genotype Taqman probe sẽ phải chứa các trình tự bắt cặp với các trình tự biến đổi và chính điều này sẽ làm cho hiệu quả bắt cặp của Taqman probe đặc hiệu cho genotype sẽ không cao, do vậy kết quả phát hiện genotype sẽ không đặc hiệu như mong muốn. Trong khi đó thiết kế probe lai sẽ dễ dàng hơn, chỉ cần quan tâm đến trình tự khác biệt cho genotype mà không nhất thiết phải quan tâm đến các biến đổi trình tự khác, vì cho dù có mang các trình tự biến đổi ngoài trình tự đặc hiệu genotype thì khi phân tích đỉnh chảy bằng chương trình HRM thì mỗi genotype sẽ biểu hiện một đỉnh chảy đặc trưng phụ thuộc chủ yếu vào trình tự đặc hiệu của genotype mà hai probe lai bắt cặp vào (xem phần probe lai). Biểu đồ 11 là một ví dụ của ứng dụng probe lai trong phát hiện genotype của HCV bằng real-time PCR đặc hiệu vùng 5’-NC.
Biểu đồ 11: Kiểu đỉnh chảy tương ứng với genotype HCV trong kết quả phân tích đỉnh chảy sản phẩm khuếch đại của real-time đặc hiệu vùng 5’NC và dùng FRET probe (trích từ Clinical Chemistry 48:12)